
分子動力学シミュレーション専用計算機 MDGRAPE-4A
私たちは、分子動力学シミュレーション用の一連の専用計算機を開発してきました。2019年には、MDGRAPE-4 [1]の改良版であるMDGRAPE-4Aが完成しました。目標の性能は、水に囲まれた典型的なタンパク質-リガンド複合体を1日あたりマイクロ秒でシミュレートできるようにすることです。これは、市販のシステムよりも1桁高速です。現在、ハードウェアシステムが完全に稼働しており、プロダクションランがシステムで実行されています。ソフトウェアの精度は、相対誤差を測定し、シミュレートされた物理的特性をGROMACS単精度の結果と比較することによって検証されました。
達成された性能は、約10万個の原子を含むシステムで1日あたり約1マイクロ秒です。MDGAPE-4Aを利用して、COVID-19の創薬プロジェクトが進行中です[2]。
MDGRAPE-4A システムアーキテクチャ
MDGRAPE-4A システムオンチップLSI
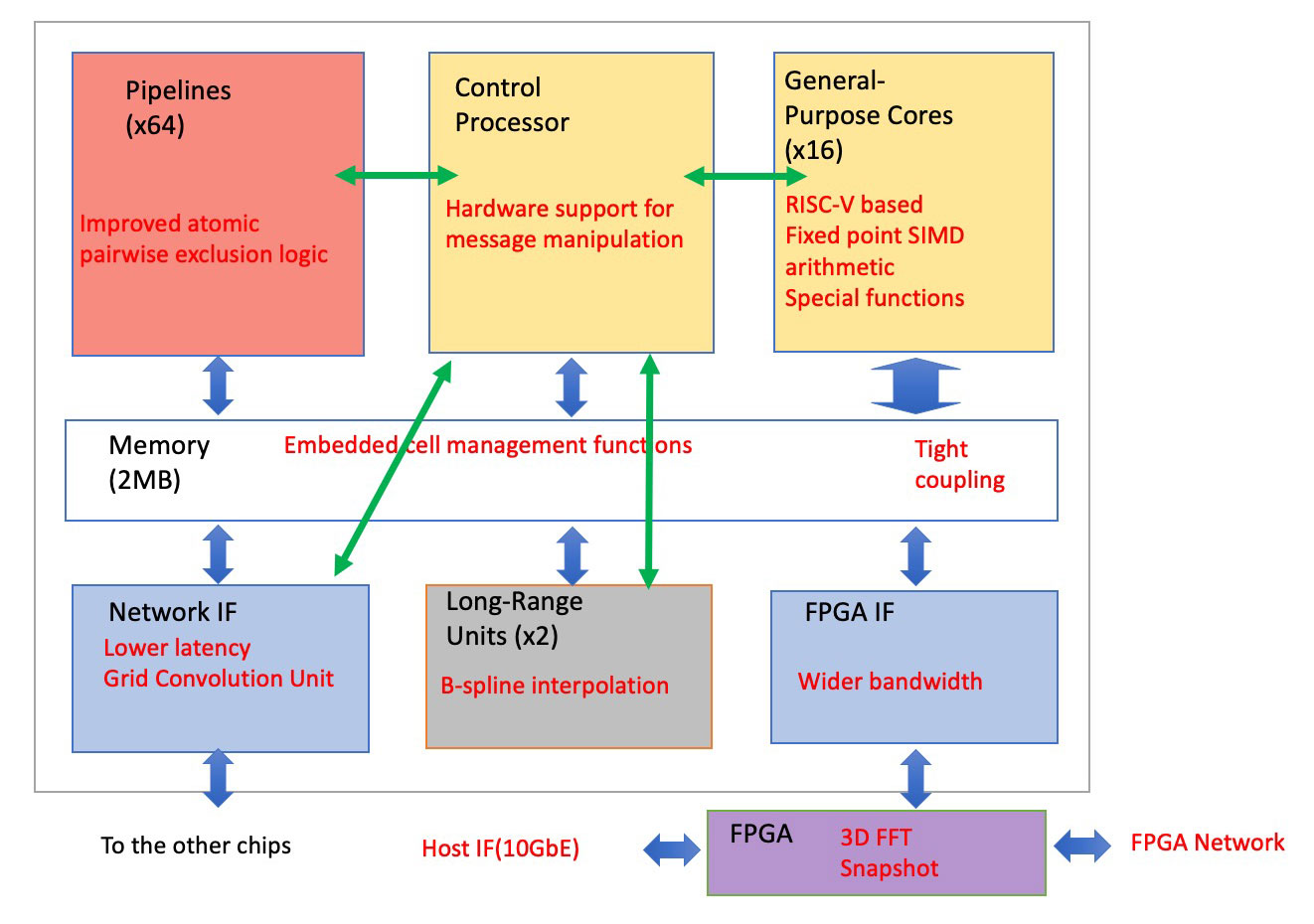
MDGRAPE-4からの改良(赤のテキスト)
Message flow (緑の矢印)
データフロー(青の矢印)
LSI設計: 理化学研究所、及び、ディー・クルー・テクノロジーズ株式会社
LSI製造:アルチップ・テクノロジーズ株式会社 TSMC 40G 16.28mm x 16.28mm
ロジック: ~5000万 ゲート
SRAM: ~72M bits
消費電力: 85W
動作クロック: 0.6GHz、0.8GHz(パイプライン)
MDGRAPE-4A ボード:8つの LSI

MDGRAPE-4A フルシステム: 64 ボード

システムの設計・製造:
東京エレクトロン デバイス株式会社への委託のもと、株式会社ホックス、インテグラン株式会社、株式会社フジクラの協力を得ました。
消費電力:65kW (推定値)
空冷 冷却システム:ヒートシンク・ファンによる空冷
開発・製造費: 約7億円(研究者の人件費を除く)
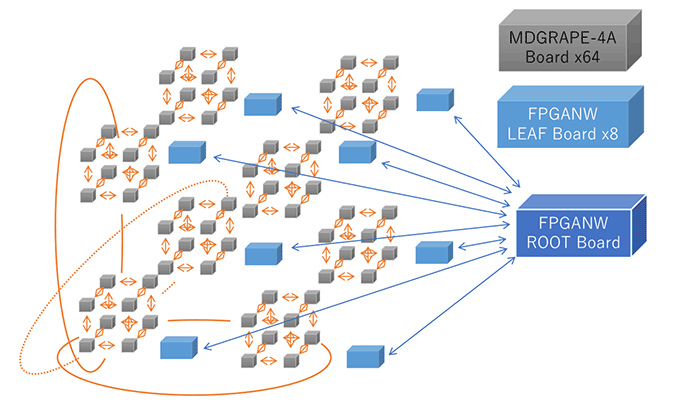
Network topology of the full system
3D-torus(8x8x8x) 6Gbps 12ch
FPGA Tree(64-8-1) 10Gbps 4ch
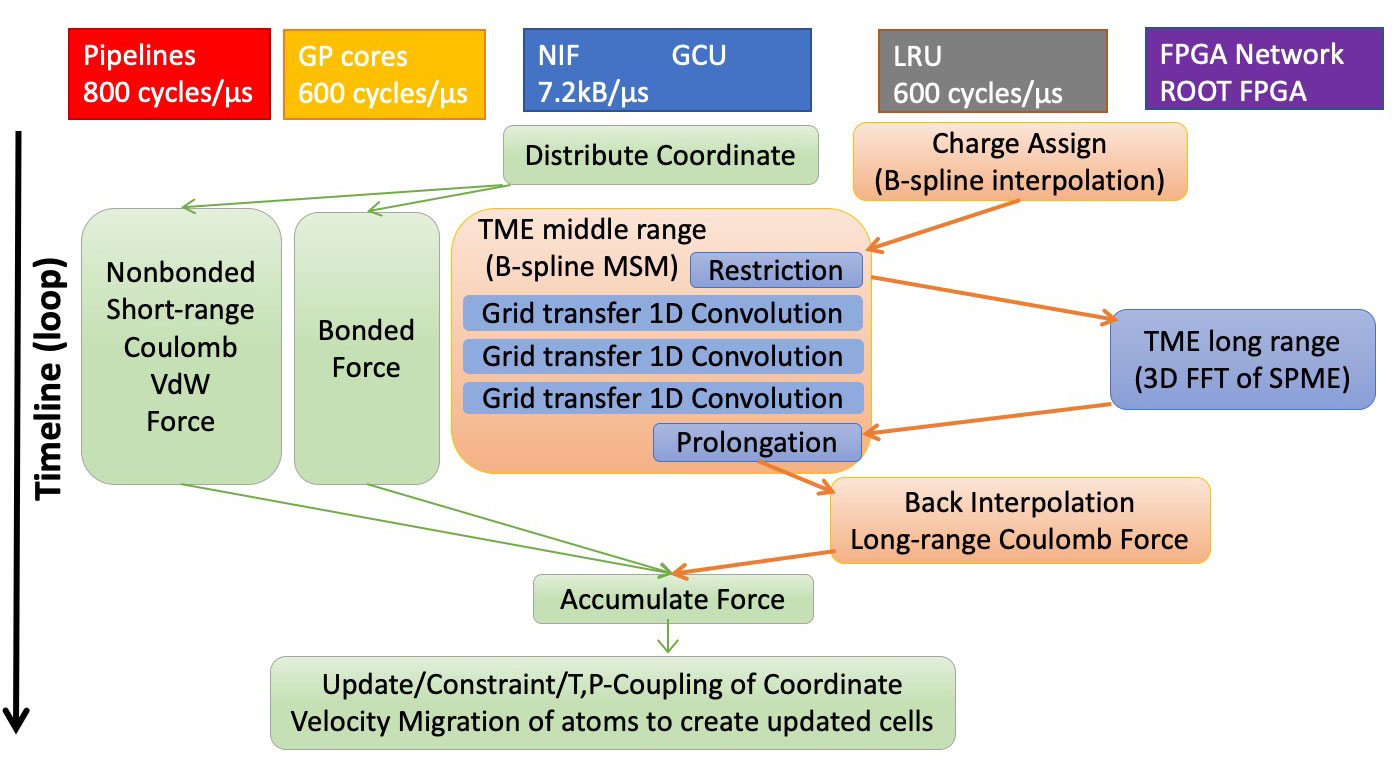
並列MD計算のシーケンス
達成されたパフォーマンス:1 MDタイムステップ(dt = 2.5fs)を完了するために200µs未満、1µs /dayを超えるシミュレーション速度。
左右にスクロールできます
| Computer System | Elapsed time for single step (µs) | Performance (µs/day, dt=2.5fs) |
|---|---|---|
| MDGRAPE-4A | 200 | 1 |
| Commodity Cluster | 1,000 | 0.2 |
| GPU | 2,000 | 0.1 |
| Laptop | 100,000 | 0.002 |
*概算値であり、詳細値はシミュレーション条件により異なります。DE Shaw Researchによって開発された専用計算機Antonは、MDGRAPE-4Aよりも1桁または2桁高速です。
ソフトウェア仕様
- GROMACS 互換の入出力コンバーター
- Velocity-Verletインテグレーター
- AMBER 力場 (CMAPなしのCHARMM、OPLS-AAは変換可能)
- TIP3P水モデルのSETTLE拘束
- 水素ボンドのRATTLE拘束
- 温度制御(速度の再スケーリング)
- 長距離静電気力 (TME)
- 圧力制御 (Monte Carlo barostat)
- より大規模で複雑なシステム(> 12万原子数、膜)の拡張
- 他のさまざまな力場の拡張 (TIP4P, CMAP)
静電相互作用のアルゴリズム
- ◆ Tensor-structured multilevel Ewald summation method (TME)[3]
- TME法は、smooth particle mesh Ewald 法(SPME)[4]および、BスプラインMSM法[5] のガウス関数による近似の組み合わせと見なすことができます。 MDGRAPE-4Aの実装では6次B-スプライン(5次の区間多項式)補間を用いたSPME法と同等の精度があります。
- ◆ Zero-Multipole Summation Method (ZMM)[6]
- 静電中和原理を利用したカットオフ法。長距離静電は計算されません。
左右にスクロールできます
| Coulomb | Performance (µs/day) |
|---|---|
| ZMM(order=2) | 1.25 |
| TME (6 order B-spline, 32 x 32 x 32 grid) | 1.12 |
システム全体のパフォーマンスを測定するためのターゲットシステム:~10nm立方体ボックスに合計100,000個の原子。システムは4096セルに分割され、各ノードは8セルを担当します。
Against COVID-19
HIV阻害剤(ネルフィナビル)のSARS-CoV-2Mproへの結合プロセスのシミュレーション
長期MDシミュレーションのアプリケーションの1つは、創薬研究です。シミュレーションの目的は、サイトポケットの形状の変化を調査し、結合経路を見つけ、サイトへのリガンドの親和性を推定することです。 (詳細は 文献[2]を参照下さい)
参考文献
- 1.Ohmura, I., et al., MDGRAPE-4: a special-purpose computer system for molecular dynamics simulations, Phil. Trans. R. Soc. A, 372:20130387, 2014.
- 2.Komatsu, T. S. et al., Drug binding dynamics of the dimeric SARS-CoV-2 main protease determined by molecular dynamics simulation, Sci. Rep. 10, 16986, 2020.
- 3.Y. M. Koyama et al., in preparation (see also Supplementary information 'Z' in [2])
- 4.Essmann, U. et al. A smooth particle mesh Ewald method. J. Chem. Phys. (1995) doi:10.1063/1.470117.
- 5.Hardy, D. J., et al., Multilevel summation with B-spline interpolation for pairwise interactions in molecular dynamics simulations, J. Chem. Phys.,144:114112, 2016.
- 6.Sakuraba, S. et al., Performance evaluation of the zero-multipole summation method in modern molecular dynamics software, J. Comp. Chem. 39 (20), 2018.
謝辞
この開発は、日本医療研究開発機構(AMED)が支援する創薬支援推進事業- 「創薬支援インフォマティックシステムの構築」の一環として実施されています。この研究は、日本学術振興会(JSPS)の支援を受けています。
LSIの設計評価にご協力いただいた大正製薬株式会社に感謝いたします。
関連リンク
- 創薬専用スパコンの開発 (プレスリリース)
- 計算分子設計研究チーム
- 共同研究・受託サービスについて
お問い合わせ先 mdgrape-contact[at]ml.riken.jp ([at]を@に変えてください)





