チームリーダー*
木川 隆則
D. Sci.
細胞構造生物学研究チーム
[2023年3月 終了]
E-mail kigawa[at]riken.jp
[at]を@に変えてください
*現:NMR維持管理・共用促進チーム 上級研究員;
生体分子動的構造研究チーム 副チームリーダー
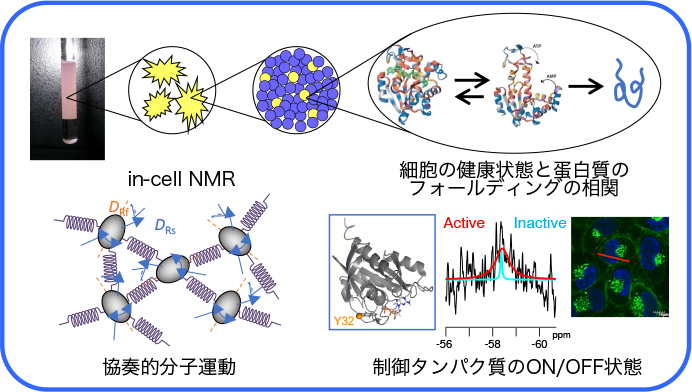
生命機能の担い手であるタンパク質などの生命分子が実際に働く場である細胞は、多種類の分子が高過密に充填されているとともに、細胞膜や細胞内小器官などにより分子の動きが制限されているなかで、様々な分子が協調して働く動的な環境となっています。近年、この細胞環境にある生命分子のかたち(立体構造)やその動的な振る舞い(動態)が、試験管内とは異なる例が見出されたことにより、細胞のなかでの分子の構造や動態を直接知ることが重要な課題となりました。本研究チームでは、主にNMR法と情報科学を駆使した解析により、細胞環境での分子構造動態を明らかにすることにより、生きた細胞における生命現象を詳細に理解することを目指しています。また、細胞環境にある分子のNMR計測は感度や分解能が著しく劣るため、これら問題を解決するための試料調製、安定同位体標識、NMR測定、およびデータ解析に関する技術の開発や高度化も進めています。さらに、得られた知見に基づいて、例えば細胞内のエネルギー産生メカニズムを活用したバイオ発電技術の開発など、生命の機能を活用した新たな技術を生み出すことにも挑戦しています。
研究テーマ
- NMR法を用いた細胞環境における生体分子の解析
- 情報科学手法を活用したNMR技術の開発
- 無細胞タンパク質合成技術を用いた安定同位体標識技術の開発
- 生体機能を活用したバイオ発電技術の開発
主要論文
Yagi H, Kasai T, Rioual E, et al.
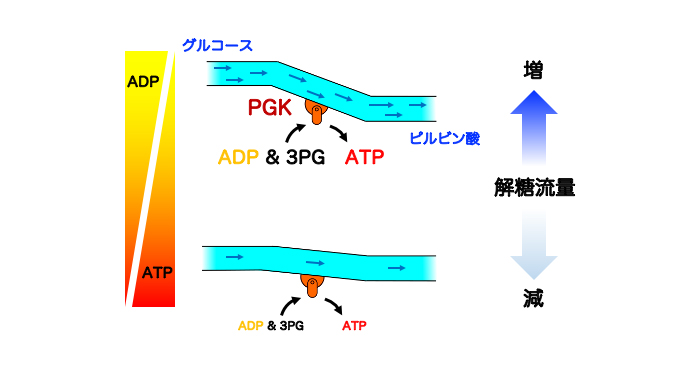
Molecular mechanism of glycolytic flux control intrinsic to human phosphoglycerate kinase.
Proceedings of the National Academy of Sciences of the United States of America
118(50), e2112986118 (2021)
doi: 10.1073/pnas.2112986118
Shitanda I, Morigayama Y, Iwashita R, et al.
Paper-based lactate biofuel cell array with high power output.
Journal of Power Sources
489, 229533 (2021)
doi: 10.1016/j.jpowsour.2021.229533
Ito K, Murayama Y, Kurokawa Y, et al.
Real-time tracking reveals catalytic roles for the two DNA binding sites of Rad51.
Nature Communications
11, 2950 (2020)
doi: 10.1038/s41467-020-16750-3
Higuchi K, Yabuki T, Ito M, Kigawa T.
Cold shock proteins improve E. coli cell-free synthesis in terms of soluble yields of aggregation-prone proteins.
Biotechnology and Bioengineering
117(6), 1628-1639 (2020)
doi: 10.1002/bit.27326
Kasai T, Ono S, Koshiba S, et al.
Amino-acid selective isotope labeling enables simultaneous overlapping signal decomposition and information extraction from NMR spectra.
Journal of Biomolecular NMR
74, 125-137 (2020)
doi: 10.1007/s10858-019-00295-9
Inomata K, Kamoshida H, Ikari M, et al.
Impact of cellular health conditions on the protein folding state in mammalian cells.
Chemical Communications (Cambridge)
53(81), 11245-11248 (2017)
doi: 10.1039/c7cc06004a
Kigawa T.
Advances in stable isotope assisted labeling strategies with information science.
Archives of Biochemistry and Biophysics
628, 17-23 (2017)
doi: 10.1016/j.abb.2017.06.014
Kasai T, Nagata K, Okada M, Kigawa T.
NMR spectral analysis using prior knowledge.
Journal of Physics: Conference Series
699(1), 012003 (2016)
doi: 10.1088/1742-6596/699/1/012003
Okamura H, Nishimura H, Nagata T, et al.
Accurate and molecular-size-tolerant NMR quantitation of diverse components in solution.
Scientific Reports
6, 21742 (2016)
doi: 10.1038/srep21742
Shigeno-Nakazawa Y, Kasai T, Ki S, et al.
A pre-metazoan origin of the CRK gene family and co-opted signaling network.
Scientific Reports
6, 34349 (2016)
doi: 10.1038/srep34349
Kasai T, Koshiba S, Yokoyama J, Kigawa T.
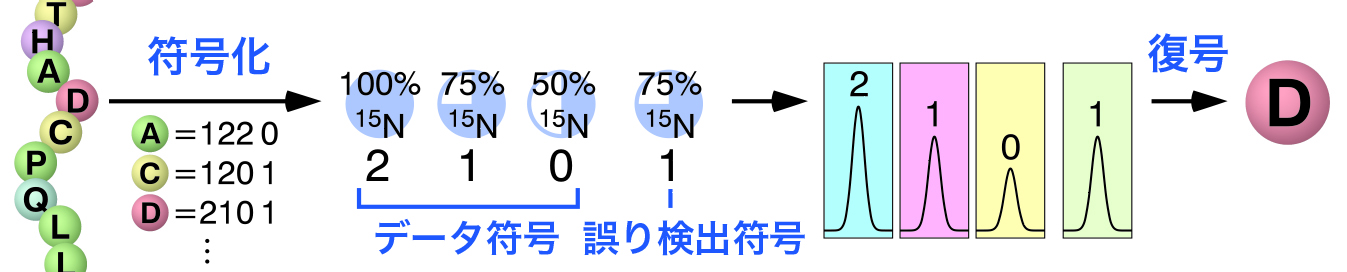
Stable isotope labeling strategy based on coding theory.
Journal of Biomolecular NMR
63(2), 213-221 (2015)
doi: 10.1007/s10858-015-9978-8
Harada R, Tochio N, Kigawa T, et al.
Reduced native state stability in crowded cellular environment due to protein-protein interactions.
Journal of the American Chemical Society
135(9), 3696-3701 (2013)
doi: 10.1021/ja3126992
Matsuda T, Furumoto S, Higuchi K, et al.
Rapid biochemical synthesis of C-11-labeled single chain variable fragment antibody for immuno-PET by cell-free protein synthesis.
Bioorganic & Medicinal Chemistry
20(22), 6579-6582 (2012)
doi: 10.1016/j.bmc.2012.09.038
Akama S, Yamamura M, Kigawa T.
A Multiphysics Model of In Vitro Transcription Coupling Enzymatic Reaction and Precipitation Formation.
Biophysical Journal
102(2), 221-230 (2012)
doi: 10.1016/j.bpj.2011.12.014
Yokoyama J, Matsuda T, Koshiba S, et al.
A practical method for cell-free protein synthesis to avoid stable isotope scrambling and dilution.
Analytical Biochemistry
411(2), 223-229 (2011)
doi: 10.1016/j.ab.2011.01.017
ニュース

2023年9月25日 BDRニュース
研究者にズームイン
汗からエネルギーを取り出す!

2022年10月27日 BDRニュース
大学院生たち
研究もスポーツも恋愛もアクティブに
2021年12月7日 研究成果
細胞がグルコース代謝量を制御する巧妙な仕組み

2021年9月10日 研究成果
細胞用電動ナノ注射器「電気浸透流ナノポンプ」を開発

2021年3月15日 研究成果
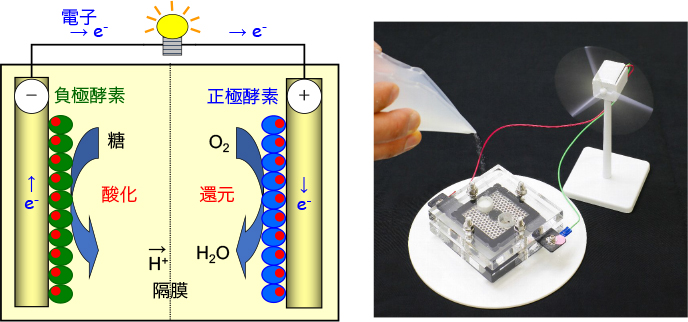
汗中乳酸から高出力を生み出す薄膜型ウェアラブルバイオ燃料電池アレイを開発

2020年10月1日 BDRニュース
BDRの研究ネホリハホリ
生化学反応の基礎と応用

2020年6月30日 研究成果
相同なDNA配列間でRad51リコンビナーゼによるDNA鎖を交換するしくみを解明
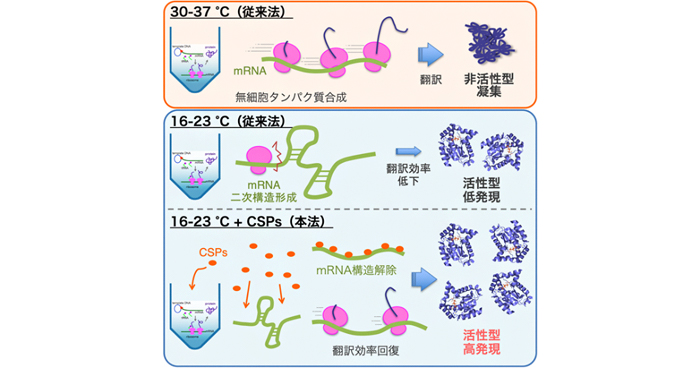
2020年4月1日 研究成果
難発現タンパク質の合成法を開発
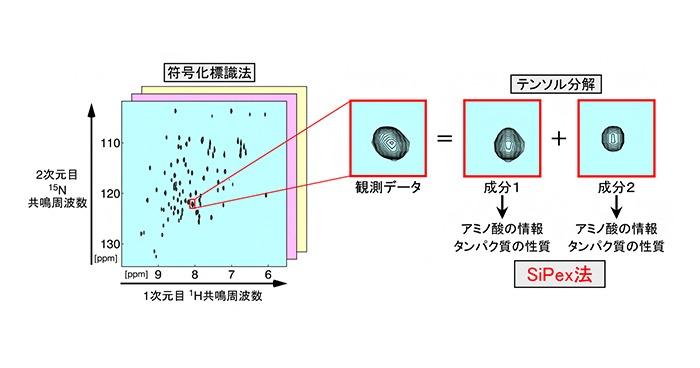
2020年1月31日 研究成果
タンパク質の構造や動きを解析する新技術を開発