チームリーダー
ZHANG Kam
Ph.D.
構造バイオインフォマティクス研究チーム
[2023年3月 終了]
E-mail kamzhang[at]riken.jp
[at]を@に変えてください
タンパク質の複雑な生物学的機能は、同じく複雑な三次元構造によって決定されます。私たちは、こうしたタンパク質の構造を計算科学的に研究することで、その機能を理解し、調節することを目指しています。そのため、タンパク質構造予測における手法を開発し、また、設計原理を応用し、新規の構造をもつ、新しい生物学的機能あるいは治療効果のあるタンパク質を創造します。X 線とCryo-EM のモデル構築と改良の新しい方法を開発し,さらには様々な創薬標的に対する新たな阻害剤の発見に向けた、計算手法の開発や応用を行います。
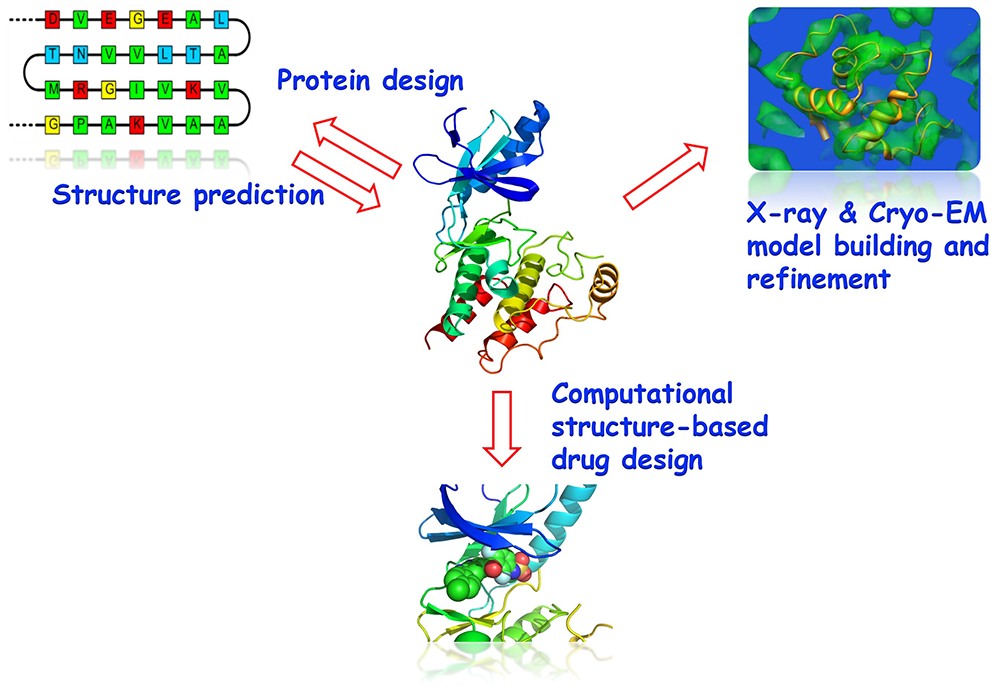
研究テーマ
- タンパク質構造予測および設計
- X線とCryo-EMモデルの構築と改良
- バーチャルスクリーニングおよび薬剤設計
主要論文
Alfi A, Popov A, Kumar A, et al.
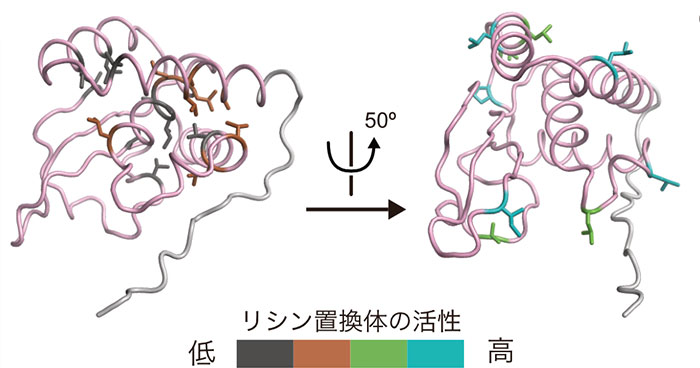
Cell-Free Mutant Analysis Combined with Structure Prediction of a Lasso Peptide Biosynthetic Protease B2.
ACS Synthetic Biology
11(6), 2022-2028 (2022)
doi: 10.1021/acssynbio.2c00176
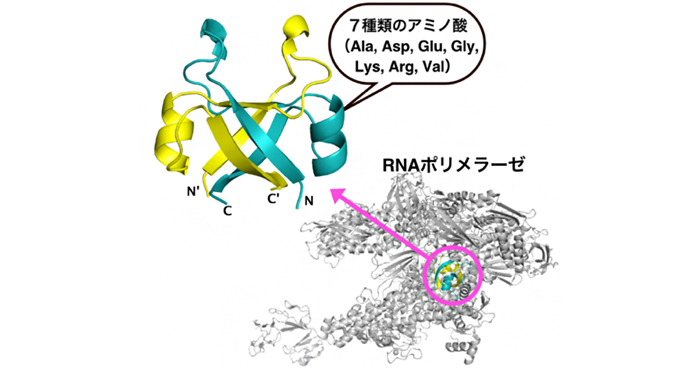
Yagi S, Padhi AK, Vucinic J, et al.
Seven Amino Acid Types Suffice to Create the Core Fold of RNA Polymerase.
Journal of the American Chemical Society
(2021)
doi: 10.1021/jacs.1c05367
Kaushik R, Zhang KYJ.
ProFitFun: A Protein Tertiary Structure Fitness Function for Quantifying the Accuracies of Model Structures.
Bioinformatics (Oxford, England)
(2021)
doi: 10.1093/bioinformatics/btab666
Padhi AK, Kumar A, Haruna KI, et al.
An integrated computational pipeline for designing high-affinity nanobodies with expanded genetic codes.
Briefings in bioinformatics
(2021)
doi: 10.1093/bib/bbab338
Jiang X, Kumar A, Motomura Y, et al.
A Series of Compounds Bearing a Dipyrido-Pyrimidine Scaffold Acting as Novel Human and Insect Pest Chitinase Inhibitors.
Journal of medicinal chemistry
63(3), 987-1001 (2020)
doi: 10.1021/acs.jmedchem.9b01154
Terada D, Voet ARD, Noguchi H, et al.
Computational design of a symmetrical ß-trefoil lectin with cancer cell binding activity.
Scientific Reports
7, 5943 (2017)
doi: 10.1038/s41598-017-06332-7
Voet ARD, Noguchi H, Addy C, et al.
Biomineralization of a Cadmium Chloride Nanocrystal by a Designed Symmetrical Protein.
Angewandte Chemie International Edition
54, 9857-9860 (2015)
doi: 10.1002/anie.201503575
Voet ARD, Noguchi H, Addy C, et al.
Computational design of a self-assembling symmetrical β-propeller protein.
Proceedings of the National Academy of Sciences of United States of America
111, 15102-15107 (2014)
doi: 10.1073/pnas.1412768111
Kumar A, Ito A, Hirohama M, et al.
Identification of Sumoylation Inhibitors Targeting a Predicted Pocket in Ubc9.
Journal of Chemical Information and Modeling
54, 2784−2793 (2014)
doi: 10.1021/ci5004015
Simoncini D, Zhang KYJ.
Efficient sampling in fragment-based protein structure prediction using an estimation of distribution algorithm.
PLoS ONE
8, e68954 (2013)
doi: 10.1371/journal.pone.0068954
Berenger F, Zhou Y, Shrestha R, Zhang KYJ.
Entropy-accelerated exact clustering of protein decoys.
Bioinformatics
27, 939 (2011)
doi: 10.1093/bioinformatics/btr072
メンバー

チームリーダーZHANG Kam
- kamzhang[at]riken.jp
([at]を@に置き換えてください)

上級研究員KUMAR Ashutosh
- akumar[at]riken.jp

研究員KAUSHIK Rahul
- rahul.kaushik[at]riken.jp

訪問研究員PADHI Aditya
- adityakumar.padhi[at]riken.jp

国際プログラム・アソシエイトTAM Chun Lai
- chunlai.tam[at]riken.jp
研修生MUHAMMAD Erma Fatiha
客員研究員KALARICKAL VIJAYAN Dileep
客員研究員BERENGER Francois
(Please replace [at] with @)