タンパク質の構造機能解析をたった2日で終わらせる
2022年6月14日
生物の体内で行われる様々な反応は、主にタンパク質が担っている。タンパク質は20種類のアミノ酸が鎖のようにつながった分子で、その鎖が特定の構造に折りたたまれて特定の機能を発揮している。したがって、生命現象を解明するためには、タンパク質の構造と機能を解析することが必須だ。ところが、タンパク質の構造機能解析はとても時間と手間のかかる作業で、順調に進んでも数ヶ月、試行錯誤にはまってしまうと数年がかりの研究になってしまう。しかし、近年開発された高性能の構造予測AIと合成生物学的手法を組み合わせることで、タンパク質の構造機能解析を驚くほど高速化できる時代が到来しつつある。
理研BDRのAlmasul Alfi研究員、Aleksandr Popov国際プログラム・アソシエイト(高機能生体分子開発チーム、田上俊輔チームリーダー)、Ashutosh Kumar上級研究員(構造バイオインフォマティクス研究チーム、Kam Zhangチームリーダー)らの国際共同研究グループ*1は、①AlphaFold2によるタンパク質の構造予測と②無細胞タンパク質合成系を利用したタンパク質の機能解析を組み合わせることで、タンパク質の構造機能解析を最短2日で完了させるプロトコルを発表した。本成果は科学誌ACS Synthetic Biologyに2022年6月8日付で掲載された。
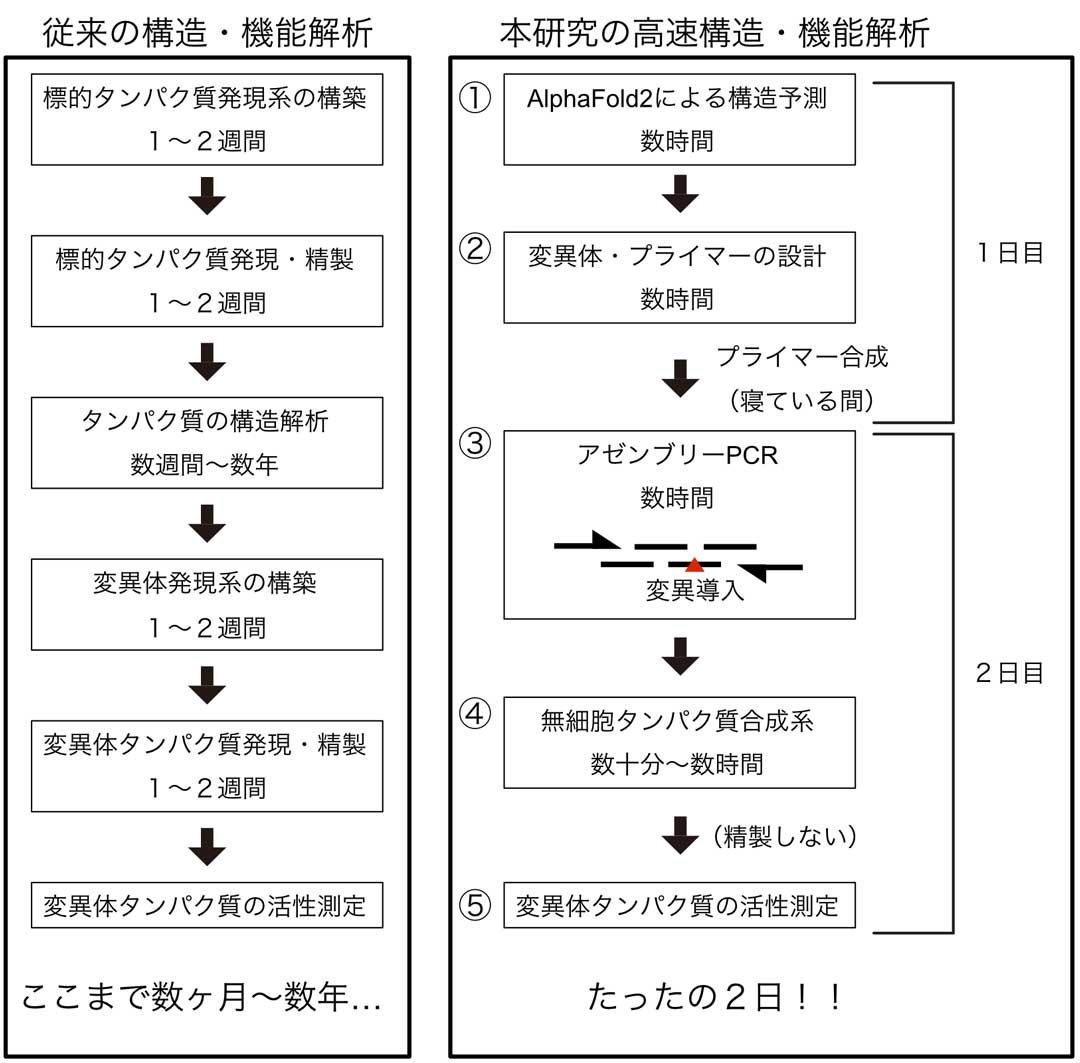
前述のように、タンパク質の構造機能解析は数ヶ月から数年かかる大変な実験である(図1左)。しかし、高性能のタンパク質構造予測AI(AlphaFold2)の登場によって、ある程度精度の高いタンパク質構造モデルがわずか数十分〜数時間で手に入るようになってきた(図1右①)。そこで、構造だけでなく機能の解析も同レベルの高速化をしたいところだ。特に、予測された構造モデルが正しいかどうかを迅速に判断するための生化学的解析プロトコルが必要とされている。
そこで、AlfiらはアゼンブリーPCRと無細胞タンパク質合成系を組み合わせた変異体解析を行い(図1右)、その結果をAlphaFold2による構造モデルと比較した。アゼンブリーPCRは短いDNA断片から目的遺伝子を組み立てる方法で(図1右③)、多くのDNA断片を反応に加える以外はほぼ通常のPCRと同じ作業で行うことができる。変異を含んだDNA断片を用いれば変異体の遺伝子を合成することもでき、通常の実験環境であれば、遺伝子の長さなどにもよるが20〜30程度の変異体遺伝子を数時間の実験で用意できる。
アゼンブリーPCRで用意した変異体遺伝子は、無細胞タンパク質合成系を用いて発現させれば、わずか数十分〜数時間で目的のタンパク質を得ることができる(図1右④)。無細胞タンパク質合成系はDNAからタンパク質を合成するために必要な酵素や基質が全て含まれた試薬で、面倒な遺伝子組換え実験(図1左)をせずに、簡単な実験でタンパク質発現を行えるという非常に大きな利点がある。このようにして得られたタンパク質変異体を精製せずにそのまま機能解析に用いることができれば(図1右⑤)、変異体のPCRから活性測定までをわずか1日で実行できる。AlphaFold2による構造予測(図1右①)や変異体作成用プライマーのデザイン・合成(図1右②)をあわせても,一連の構造機能解析が合計2日で終わることになる。なお、このようなタンパク質の無精製実験を行う際には、PUREシステムのようなコンポーネントがきちんと分かっている無細胞タンパク質合成キットを用いることが望ましい。
このプロトコルを用いて、Alfiらは実際にlassoペプチドという特殊な構造化ペプチド合成系の解析を行った(図2A)。lassoペプチドはまず直鎖状の前駆体ペプチドとして合成され、B1タンパク質によって認識された後に、B2タンパク質による切断を受ける。さらにCタンパク質による環状化を受けて最終的なlassoペプチドの構造ができあがる。これまでにB1タンパク質と前駆体ペプチドの複合体の構造解析には成功していたが(Sumida et al., ACS Chemical Biology, 2019)、B2タンパク質やCタンパク質は可溶性が低く、構造解析ができていなかった。そこで、本研究では、B2タンパク質の構造機能解析をおこなうため、無細胞タンパク質合成系を利用したB2タンパク質の変異体活性測定を行い、その結果をAlphaFold2によるB2タンパク質の構造予測と比較した。
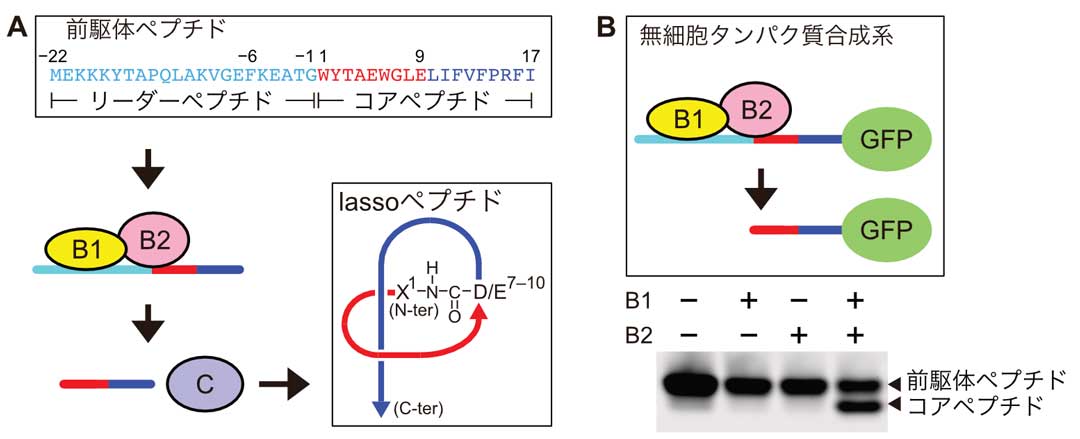
図2. (A)lassoペプチド合成系と(B)無細胞タンパク質合成系をもちいたB2タンパク質の活性
蛍光タンパク質(GFP)と連結した前駆体ペプチドをB1・B2タンパク質とともに無細胞タンパク質合成系で発現させ、タンパク質発現と同時に前駆体ペプチドの切断反応を行うB2タンパク質の活性測定系(図2B)を構築した。この実験系をもちいて、B2タンパク質に含まれる疎水性アミノ酸残基のいずれかを親水性のリシンに置換した34種類の変異体の活性測定を行い、その結果をAlphaFold2による構造モデル上にマップした(図3)。タンパク質は疎水性アミノ酸を中心に集めて『疎水性コア』をつくることでその立体構造を形成するが、リシンのような荷電アミノ酸を疎水性コアに導入するとタンパク質構造が崩れてしまう。AlphaFold2によって予測されたB2タンパク質の疎水性コアにリシンを導入した場合はB2タンパク質の活性が失われたのに対して(図3A)、リシンに置換しても活性を失わない部位は全てタンパク質表面上にあった(図3B)。このことから、AlphaFold2が高い精度でB2タンパク質の構造を予測できていることが確認できた。さらに、表面近くにあるにも関わらずリシン置換によって活性を失った部位は、システインプロテアーゼ特有のCys-His-Glu構造がある活性部位の近傍(図3C)、もしくはB1タンパク質との結合部位であると予測された(図3D、複合体の構造予測については論文を参照)。したがって,非常に簡単な実験で、簡易的にではあるが、①構造予測の信用性の確認、②活性部位の確認、③複合体形成表面の確認を同時に行うことができた。
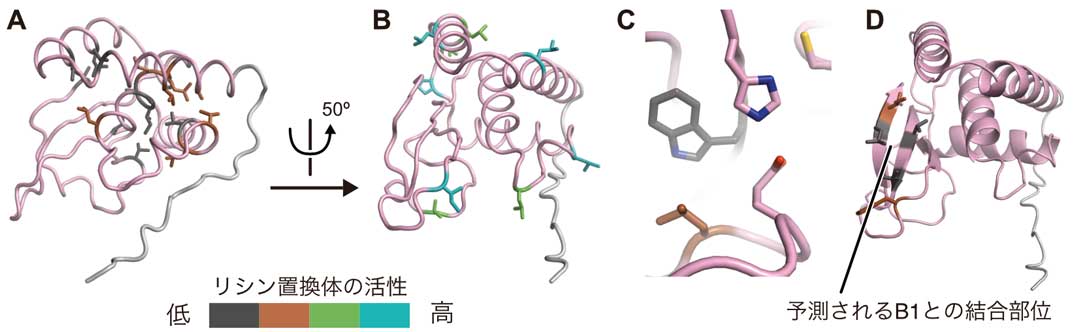
図3.lassoペプチド合成系B2タンパク質の予測構造と変異体解析の結果(論文より引用)
「このような高速構造機能解析・変異体解析手法は、他の多くのタンパク質に利用可能であり、今後様々なタンパク質の研究に応用できます。」と田上チームリーダー。実際の実験は、目的遺伝子のサイズや機能によって2段階のアゼンブリーPCRなどが必要になる場合もある。特に無細胞タンパク質合成系をもちいたタンパク質活性測定系については、それぞれの目的タンパク質についてアッセイ系を工夫する必要があるが、一度実験系を立ち上げてしまえば追加の変異体の解析なども迅速に行うことができるため、非常に便利な手法として利用できるだろう。
高橋 涼香(BDR広報グループ)
- *1 本研究は以下の研究グループの国際共同研究で実施されました。
- Almasul Alfi研究員(理化学研究所生命機能科学研究センター高機能生体分子開発チーム)
- Aleksandr Popov国際プログラム・アソシエイト(理化学研究所生命機能科学研究センター高機能生体分子開発チーム、スコルコボ科学技術大学)
- Ashutosh Kumar上級研究員(構造バイオインフォマティクス研究チーム)
- Kam Zhangチームリーダー(構造バイオインフォマティクス研究チーム)
- Svetlana Dubiley研究室長(スコルコボ科学技術大学、ロシア科学アカデミー遺伝生物学研究所)
- Konstantin Severinov教授(スコルコボ科学技術大学、ロシア科学アカデミー遺伝生物学研究所、ワクスマン微生物学研究所)
- 田上俊輔チームリーダー(理化学研究所生命機能科学研究センター高機能生体分子開発チーム)
関連リンク
掲載された論文





