
チームリーダー
杉田 有治
D.Sci.
分子機能シミュレーション研究チーム
[2025年3月 BDR兼務終了(本務:理研開拓研究所)]
E-mail sugita@riken.jp
当チームでは、「富岳」などのスパコンを用いた分子動力学シミュレーションを行い、蛋白質や核酸、糖鎖、脂質膜など生体分子の構造・ダイナミクス・機能の関係を解明します。分子動力学シミュレーションから自由エネルギー変化を求める様々な手法を開発し、分子動力学ソフトウェアGENESISに導入・公開します。これにより、基礎科学研究だけでなく、創薬応用計算にも貢献していきます。
研究テーマ
- 細胞内分子混雑環境でのタンパク質と水の構造とダイナミクス
- タンパク質大規模構造変化のシミュレーションと実験データの解析
- ブラウン運動シミュレーションを用いた長時間ダイナミクス
主要論文
Chong SH, Oshima H, Sugita Y.
Allosteric changes in the conformational landscape of Src kinase upon substrate binding.
Journal of Molecular Biology
(2024)
doi: 10.1016/j.jmb.2024.168871
Jung J, Yagi K, Tan C, et al.
GENESIS 2.1: High-Performance Molecular Dynamics Software for Enhanced Sampling and Free-Energy Calculations for Atomistic, Coarse-Grained, and Quantum Mechanics/Molecular Mechanics Models.
Journal of Physical Chemistry. B
128(25), 6028-6048 (2024)
doi: 10.1021/acs.jpcb.4c02096
Chyży P, Kulik M, Shinobu A, et al.
Molecular dynamics in multidimensional space explains how mutations affect the association path of neomycin to a riboswitch.
Proceedings of the National Academy of Sciences of the United States of America
121(15), e2317197121 (2024)
doi: 10.1073/pnas.2317197121
Shinobu A, Re S, Sugita Y.
Practical Protocols for Efficient Sampling of Kinase-Inhibitor Binding Pathways Using Two-Dimensional Replica-Exchange Molecular Dynamics.
Frontiers in Molecular Biosciences
9, 878830 (2022)
doi: 10.3389/fmolb.2022.878830
Matsubara D, Kasahara K, Dokainish HM, et al.
Modified Protein-Water Interactions in CHARMM36m for Thermodynamics and Kinetics of Proteins in Dilute and Crowded Solutions.
Molecules
27(17), 5726 (2022)
doi: 10.3390/molecules27175726
Oshima H, Sugita Y.
Modified Hamiltonian in FEP Calculations for Reducing the Computational Cost of Electrostatic Interactions
Journal of Chemical Information and Modeling
62(11), 2846-2856 (2022)
doi: 10.1021/acs.jcim.1c01532
Fujiyama K, Kato N, Re S, et al.
Molecular Basis for Two Stereoselective Diels-Alderases that Produce Decalin Skeletons*.
Angewandte Chemie
60(41), 22401-22410 (2021)
doi: 10.1002/anie.202106186
Kasahara K, Re S, Nawrocki G, et al.
Reduced efficacy of a Src kinase inhibitor in crowded protein solution.
Nature Communications
12(1), 4099 (2021)
doi: 10.1038/s41467-021-24349-5
Shinobu A, Kobayashi C, Matsunaga Y, Sugita Y.
Coarse-Grained Modeling of Multiple Pathways in Conformational Transitions of Multi-Domain Proteins.
Journal of Chemical Information and Modeling
61(5), 2427-2443 (2021)
doi: 10.1021/acs.jcim.1c00286
Re S, Oshima H, Kasahara K, et al.
Encounter Complexes and Hidden Poses of Kinase-Inhibitor Binding on the Free-Energy Landscape.
Proceedings of the National Academy of Sciences of the United States of America
116, 18404-18409 (2019)
doi: 10.1073/pnas.1904707116
メンバー
ニュース

2024年1月18日 研究成果
ウイルスのスパイクタンパク質でウミホタルの発光基質が発光
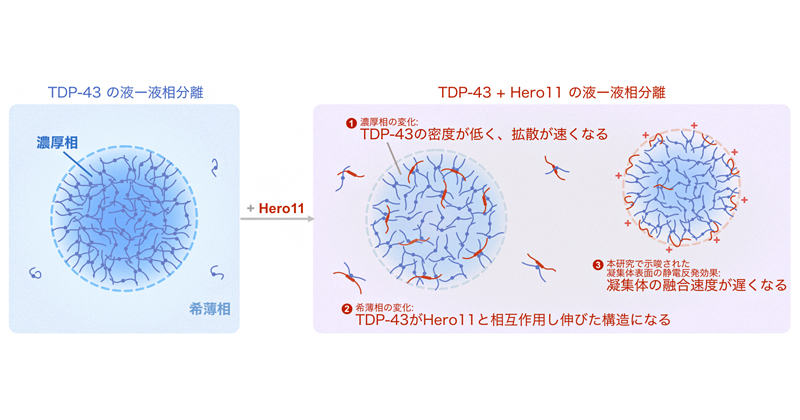
2023年3月3日 研究成果
タンパク質の凝縮を制御する分子機構
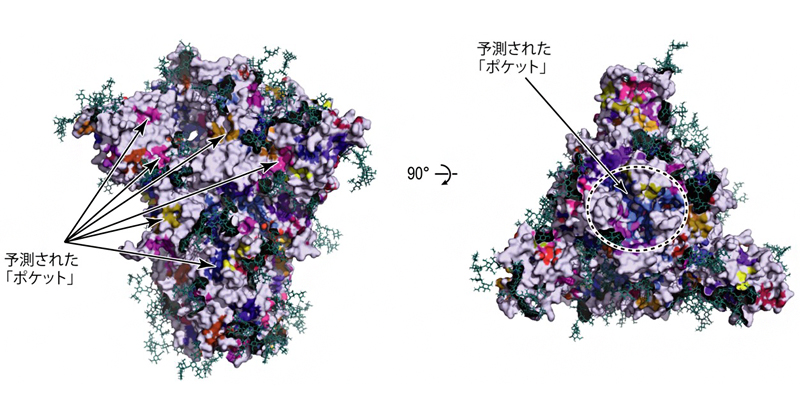
2022年4月25日 研究成果
スパイクタンパク質の構造変化を予測

2022年4月15日 BDRニュース
文部科学大臣表彰の受賞について

2022年3月23日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について

2021年7月30日 BDRニュース
国立大学法人信州大学との教育・研究に関する連携協定について
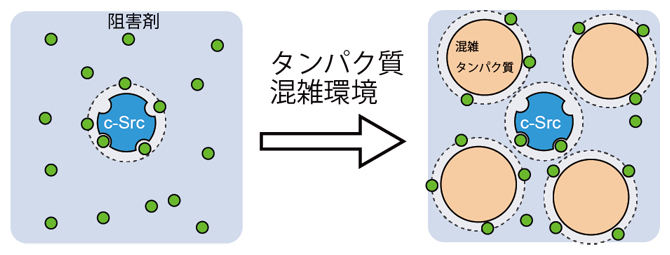
2021年7月9日 研究成果
混雑した細胞内で薬はどう効くのか

2021年5月7日 BDRニュース
BDRの研究ネホリハホリ
それは一瞬のできごと
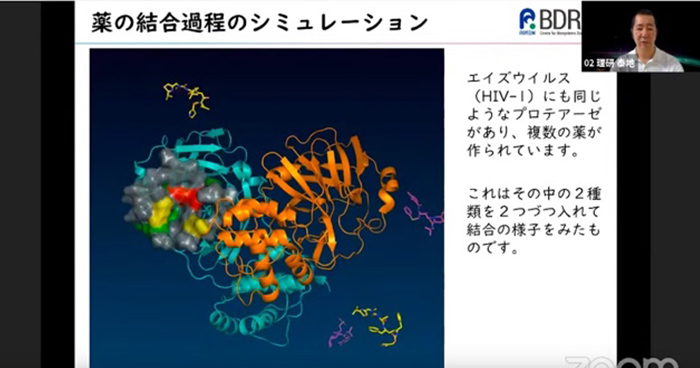
2020年8月21日 BDRニュース
一般向けイベント
中高生のための夏の特別授業「新型コロナウイルスに挑む生命科学・計算科学」を開催

2020年2月5日 研究成果
研究最前線:GENESISで創薬が変わる(杉田有治チームリーダー)[PDF 4.5MB]

2019年11月23日 BDRニュース
李 秀栄 上級研究員(分子機能シミュレーション研究チーム)のエッセイが産経新聞の連載「科学の中身」に掲載されました
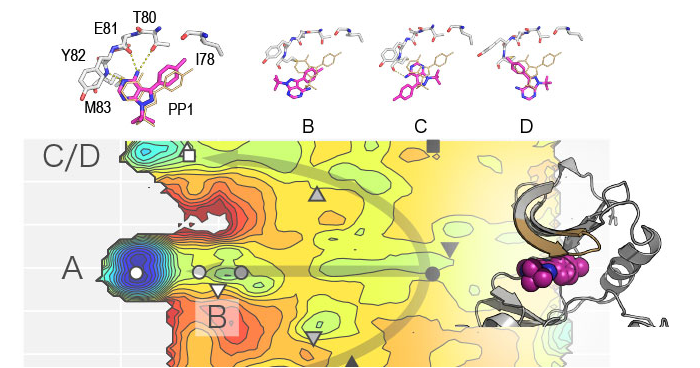
2019年9月6日 研究成果
酵素-阻害剤結合の初期会合体を予測





