
光学顕微鏡を用いた蛋白質1分子動態観察と次世代シークエンサ関連の手法開発の経験を基に、バイオイメージング・シークエンシング・機械学習を組み合わせた新規1細胞計測法を開発し、三次元空間計測へも展開しています。DNA分子バーコード法の開発により、高精度にRNA 分子を定量できるDigital RNA sequencing (dRNA-seq)を実現し、また、細菌の種類を高精度で区別して1細胞レベルで定量できる、新しい細菌叢解析法も開発しました。これらの技術開発を基盤として、生命システムの理解や、幹細胞・オルガノイドを用いた研究などにより医科学への貢献を目指しています。免疫に関わる細胞などを対象に、たくさんの共同研究も行っています。
研究テーマ
- 幹細胞・オルガノイド
- 免疫細胞
- 空間細胞計測
- 細菌叢解析
- DNA分子バーコード法
主要論文
OkaniwaT, Kryukov K, Shiroguchi K.
Finding differentially expressed genes between cell fates predicted by image-based deep learning
Biophysics and Physicobiology
22(4), e220022 (2025)
doi: 10.2142/biophysico.bppb-v22.0022
Wataru Ise, Takuya Koike, Nozomi Shimada , et al.
KLF2 expression in IgG plasma cells at their induction site regulates the migration program
J Exp Med
222 (5):e20241019, (2025)
doi: 10.1084/jem.20241019
Kanaya T, Jinnohara T, Sakakibara S, et al.
RelB and C/EBPα critically regulate the development of Peyer’s patch mononuclear phagocytes
Mucosal Immunology
(2024)
doi: 10.1016/j.mucimm.2024.10.005
Jin J, Liu X, Shiroguchi K.
Long journey of 16S rRNA-amplicon sequencing toward cell-based functional bacterial microbiota characterization
iMetaOmics
(2024)
doi: 10.1002/imo2.9
Nakai-Futatsugi Y, Jin J, Ogawa T, et al.
Pigmentation level of human iPSC-derived retinal pigment epithelium cell does not indicate a specific gene expression profile
eLife
12, RP92510 (2024)
doi: 10.7554/eLife.92510
Takahashi S, Ochiai S, Jin J, et al.
Sensory neuronal STAT3 is critical for IL-31 receptor expression and inflammatory itch
Cell Reports
(2023)
doi: /10.1016/j.celrep.2023.113433
Jin J, Yamamoto R, Shiroguchi K, et al.
High-throughput identification and quantification of bacterial cells in the microbiota based on 16S rRNA sequencing with single-base accuracy using BarBIQ
Nature Protocols
(2023)
doi: /10.1038/s41596-023-00906-8
Cui G, Shimba A, Jin J, et al.
CD45 Alleviates Airway Inflammation and Lung Fibrosis by Limiting Expansion and Activation of ILC2s.
Proceedings of the National Academy of Sciences of the United States of America
120(36), e2215941120 (2023)
doi: 10.1073/pnas.2215941120
Fujimura T, Enomoto Y, Katsura H, et al.
Identifying a Lung Stem Cell Subpopulation by Combining Single-Cell Morphometrics, Organoid Culture, and Transcriptomics.
Stem Cells
41(8), 809-820 (2023)
doi: 10.1093/stmcls/sxad044
Jin J, Ogawa T, Hojo N, et al.
Robotic data acquisition with deep learning enables cell image-based prediction of transcriptomic phenotypes.
Proceedings of the National Academy of Sciences of the United States of America
120(1), e2210283120 (2023)
doi: 10.1073/pnas.2210283120
Cui G, Shimba A, Jin J, et al.
A circulating subset of iNKT cells mediates antitumor and antiviral immunity.
Science Immunology
7(76), eabj8760 (2022)
doi: 10.1126/sciimmunol.abj8760
Jin J, Yamamoto R, Takeuchi T, et al.
High-throughput identification and quantification of single bacterial cells in the microbiota.
Nature Communications
13, 863 (2022)
doi: 10.1038/s41467-022-28426-1
Zhang B, Vogelzang A, Miyajima M, et al.
B cell-derived GABA elicits IL-10+ macrophages to limit anti-tumour immunity.
Nature
599(7885), 471-476 (2021)
doi: 10.1038/s41586-021-04082-1
Nishida M, Yamashita N, Ogawa T, et al.
Mitochondrial reactive oxygen species trigger metformin-dependent antitumor immunity via activation of Nrf2/mTORC1/p62 axis in tumor-infiltrating CD8T lymphocytes.
Journal for immunotherapy of cancer
9, e002954 (2021)
doi: 10.1136/jitc-2021-002954
ニュース
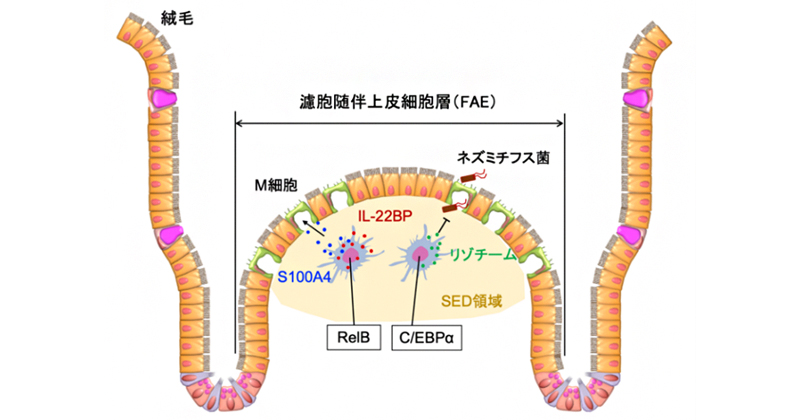
2024年11月12日 研究成果
パイエル板免疫細胞の機能調節機構を解明
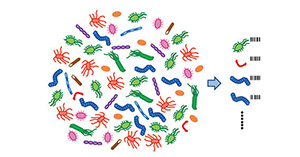
2024年7月19日 研究成果
マーカー遺伝子を用いた細菌叢解析法のおよそ50年の歴史を俯瞰

2024年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について
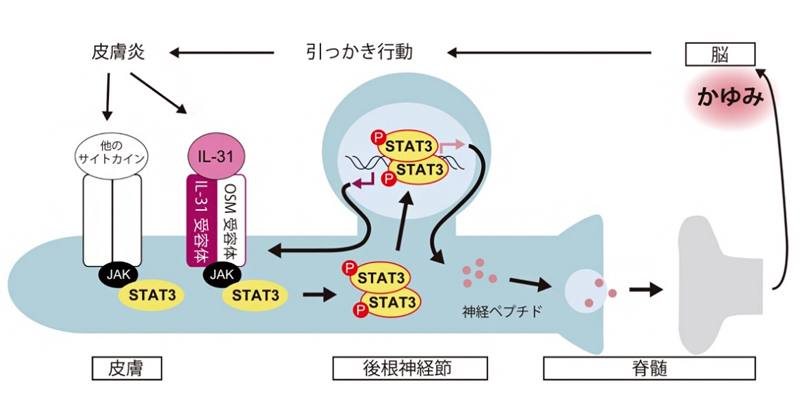
2023年11月29日 研究成果
アトピー性皮膚炎のかゆみ伝達機序を解明

2023年11月28日 研究成果
細胞単位で計測できる新しい細菌叢解析法のプロトコルを公開
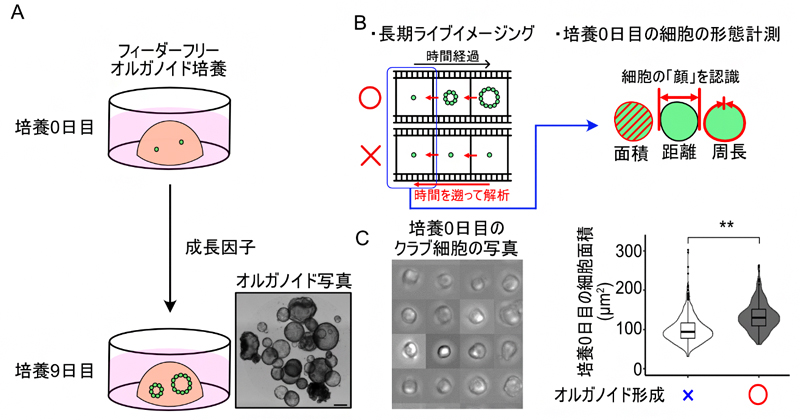
2023年7月20日 研究成果
肺に存在する新たな幹細胞の発見

2023年1月17日 研究成果
プレスリリース解説 vol.16「単一細胞の“顔つき”から“調子”を見抜く」
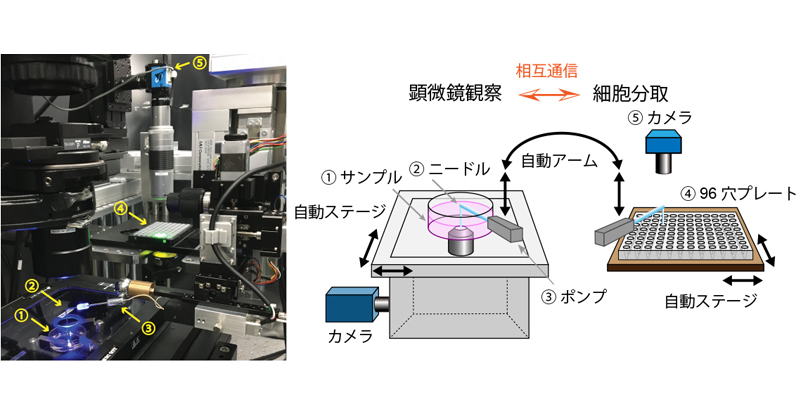
2022年12月27日 研究成果
細胞一つずつの“顔つき”から“調子”を見抜く
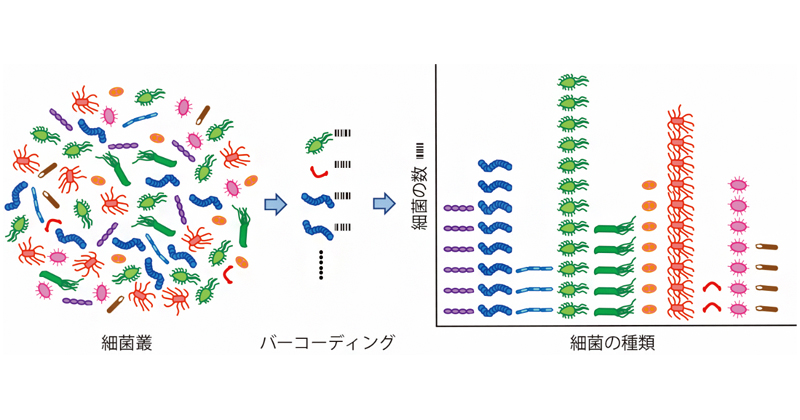
2022年2月22日 研究成果
細菌一つを見分ける細菌叢計測法の開発
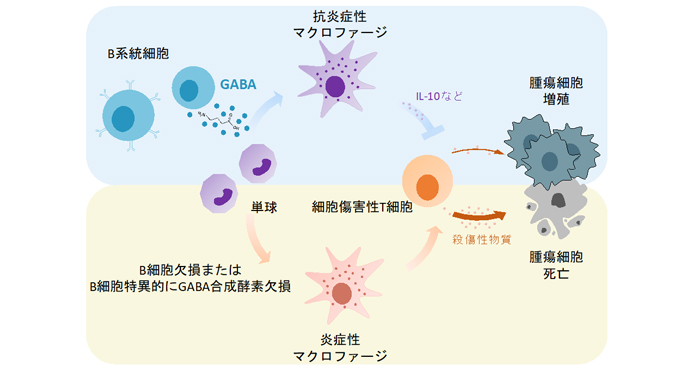
2021年11月10日 研究成果
GABAを標的とする抗腫瘍免疫機構

2020年2月4日 BDRニュース
城口克之チームリーダーが中谷賞奨励賞を受賞
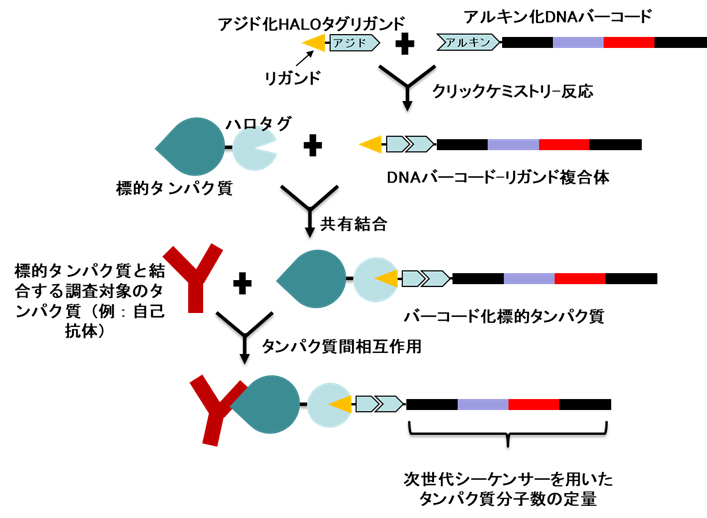
2019年11月29日 研究成果
DNAバーコーディングによるタンパク質のデジタル定量法






