チームリーダー
坂本 健作
Ph.D.
非天然型アミノ酸技術研究チーム
[2023年3月 終了]
E-mail kensaku.sakamoto[at]riken.jp
[at]を@に変えてください
医療や生命科学研究に有用なタンパク質を合成する方法として、生きた細胞にタンパク質の設計図を読みこませて、細胞に作ってもらうことができます。本来なら生き物が使わない新規なアミノ酸をも使えるように改変した細胞を用いると、目的のタンパク質に目印をつけたり、薬剤と連結したりすることが容易になります。また、これまでにない新しいタンパク質の形や働きを実現することができるかもしれません。新規アミノ酸のような人工のコンポーネントを含んだタンパク 質の生産技術を開発し、創薬に対してもユニークで最先端の技術支援を行います。
合成生物学とは?
現在流行りの「合成生物学(Synthetic Biology)」ですが、これと言って決まった定義はありません。私たちは、その有用性に着目し、従来の天然資源に依存した発見から、人為的に作成した多様性の中から新しいものを見出すことへとアプローチが変化している現状が、合成生物学の背景にあると考えています。ここでは、そのコンセプトと、生物の各階層における多様性の基盤についてまとめてみました(図1)。

図1
タンパク質の部位特異的修飾法
新規アミノ酸をタンパク質の望みの部位に自在に導入する私たちの技術を使って、タンパク質の修飾が簡単になりました。今後、バイオ医薬品はタンパク質そのままではなく、手を加えた状態(修飾体)で効用をアップすることが望まれます。ここでは、昆虫細胞を遺伝的に改変して新規アミノ酸(AzF)の導入を可能にしています(下図)。
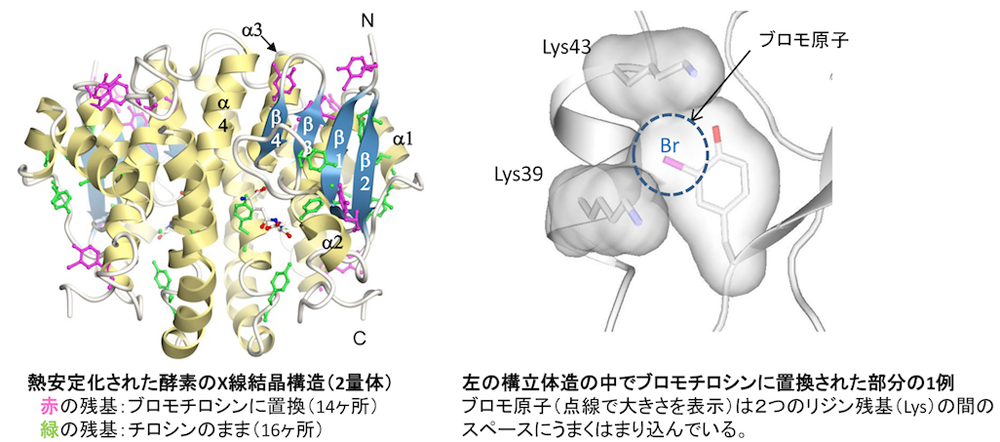
タンパク質の構造安定化技術:非天然のアミノ酸(ここではブロモチロシン)を酵素に組込むことで構造の安定化が実現できます。左の図には安定化された酵素の立体構造を実際にX線結晶構造解析技術によって明らかにしたものを示しています。右の図では、その中のあるブロモチロシンが、リジン残基の間にもともと存在していたスペースにうまくはまりこんで酵素を安定化していることがわかりました。他に組込まれたブロモチロシンも同じような働きをしていることがわかっています。
研究テーマ
- 生細胞における遺伝暗号の改変
- 非天然型アミノ酸の導入による新規タンパク質の創出
- 医薬品開発につながる抗体のエンジニアリング
主要論文
Wada A, Umeki Y, Annoura T, Saito-Nakano Y.
In Vitro and In Vivo Antiamebic Activity of Iron-Targeting Polypyridine Compounds against Enteric Protozoan Parasite Entamoeba histolytica.
ACS Infectious Diseases
8(3), 457-462 (2022)
doi: 10.1021/acsinfecdis.1c00418
Padhi AK, Kumar A, Haruna KI, et al.
An integrated computational pipeline for designing high-affinity nanobodies with expanded genetic codes.
Briefings in Bioinformatics
22(6), bbab338 (2021)
doi: 10.1093/bib/bbab338
Terasawa K, Kato Y, Ikami Y, et al.
Direct homophilic interaction of LAMP2A with the two-domain architecture revealed by site-directed photo-crosslinks and steric hindrances in mammalian cells.
Autophagy
(2021)
doi: 10.1080/15548627.2021.1911017
Hayashi A, Haruna KI, Sato H, et al.
Incorporation of Halogenated Amino Acids into Antibody Fragments at Multiple Specific Sites Enhances Antigen Binding.
Chembiochem
22(1), 120-123 (2021)
doi: 10.1002/cbic.202000429
Seki E, Yanagisawa T, Kuratani M, et al.
Fully Productive Cell-Free Genetic Code Expansion by Structure-Based Engineering of Methanomethylophilus alvus Pyrrolysyl-tRNA Synthetase.
Acs Synthetic Biology
9(4), 718-732 (2020)
doi: 10.1021/acssynbio.9b00288
Sakamoto K, Hayashi A.
Synthetic tyrosine tRNA molecules with noncanonical secondary structures.
International Journal of Molecular Sciences
20(1), 92 (2019)
doi: 10.3390/ijms20010092
Yanagisawa T, Kuratani M, Seki E, et al.
Structural Basis for Genetic-Code Expansion with Bulky Lysine Derivatives by an Engineered Pyrrolysyl-tRNA Synthetase.
Cell chemical biology
26(7), 936-949 (2019)
doi: 10.1016/j.chembiol.2019.03.008
Teramoto H, Amano Y, Iraha F, et al.
Genetic code expansion of the silkworm Bombyx mori to functionalize silk fiber.
ACS Synthetic Biology
7, 801-806 (2018)
doi: 10.1021/acssynbio.7b00437
Ohtake K, Mukai T, Iraha F, et al.
Engineering an automaturing transglutaminase with enhanced thermostability by genetic code expansion with two codon reassignments.
ACS Synthetic Biology
7, 2170-2176 (2018)
doi: 10.1021/acssynbio.8b00157
Handoko L, Kaczkowski B, Hon C-C, et al.
JQ1 affects BRD2-dependent and independent transcription regulation without disrupting H4-hyperacetylated chromatin states.
Epigenetics
13, 410-431 (2018)
doi: 10.1080/15592294.2018.1469891
Yamaguchi A, Iraha F, Ohtake K, Sakamoto K.
Pyrrolysyl-tRNA Synthetase with a Unique Architecture Enhances the Availability of Lysine Derivatives in Synthetic Genetic Codes.
Molecules (Basel, Switzerland)
23(10), E2460 (2018)
doi: 10.3390/molecules23102460
メンバー

チームリーダー坂本 健作
- kensaku.sakamoto[at]riken.jp
([at]を@に置き換えてください)
副チームリーダー脇山 素明
- motoaki.wakiyama@riken.jp
専任研究員和田 章
- awada[at]riken.jp
上級技師松元(林) 明子
- akiko.matsumoto[at]riken.jp
研究員大竹 和正
- kazumasa.ohtake[at]riken.jp
技師高橋 美穂子
- mihoko.takahashi[at]riken.jp
(Please replace [at] with @)