発生現象は、分子、細胞、組織という異なる空間スケールでの現象からなるマルチスケールなシステムであると同時に、分子による時空間情報の伝達や読み取り、細胞の増殖や運動を通じた組織内応力の発生、それに伴う幾何学的な組織変形といったマルチフィジクスなシステムでもあります。本研究チームでは、理論と実験の両研究を通じ、異なるスケールや性質をもつ現象、及び現象間の動的かつ協調的な相互作用を定量的に理解することを目指します。
研究テーマ
- 器官レベルの形態形成定量イメージングと幾何学的解析
- 成長組織内における細胞の時空間認識メカニズム
- 器官形態形成過程の力学モデリング
- 細胞集団運動を通じた空間パターニングの計測とモデリング
主要論文
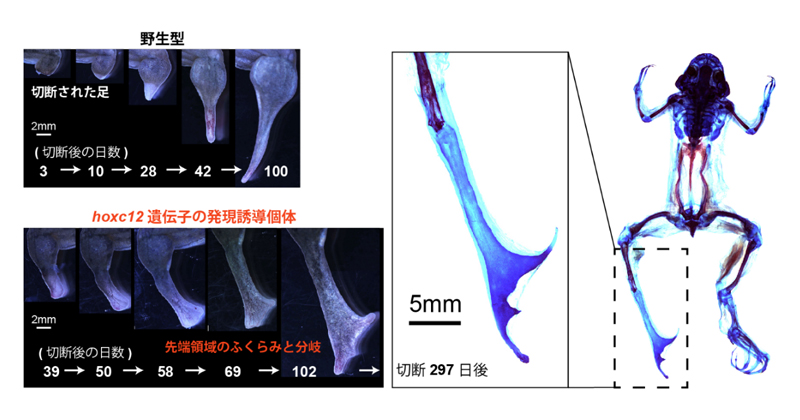
Kawasumi-Kita A, Lee SW, Ohtsuka D, et al.
hoxc12/c13 as key regulators for rebooting the developmental program in Xenopus limb regeneration.
Nature Communications
15(1), 3340 (2024)
doi: 10.1038/s41467-024-47093-y
Morishita Y, Lee SW, Suzuki T, et al.
An archetype and scaling of developmental tissue dynamics across species.
Nature Communications
14(1), 8199 (2023)
doi: 10.1038/s41467-023-43902-y
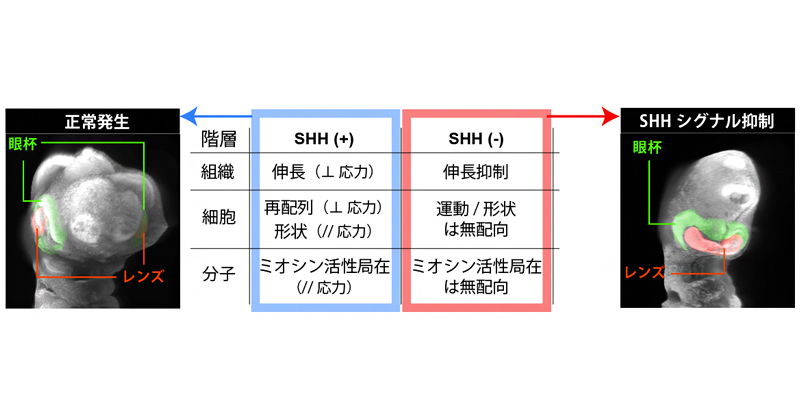
Ohtsuka D, Kida N, Lee SW, et al.
Cell disorientation by loss of SHH-dependent mechanosensation causes cyclopia.
Science Advances
8(28), eabn2330 (2022)
doi: 10.1126/sciadv.abn2330
Kawahira N, Ohtsuka D, Kida N, et al.
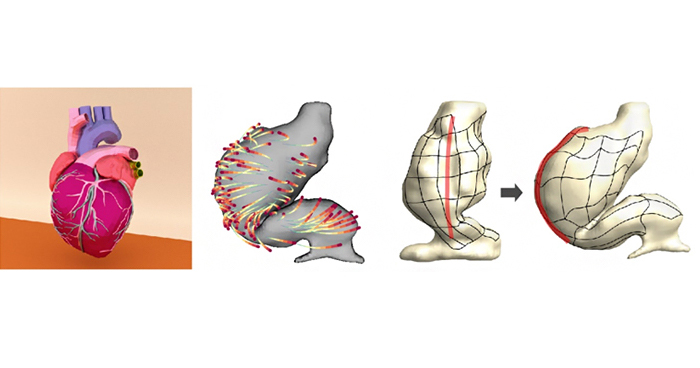
Quantitative analysis of 3D tissue deformation reveals key cellular mechanism associated with initial heart looping.
Cell Reports
30, 3889-3903 (2020)
doi: 10.1016/j.celrep.2020.02.071
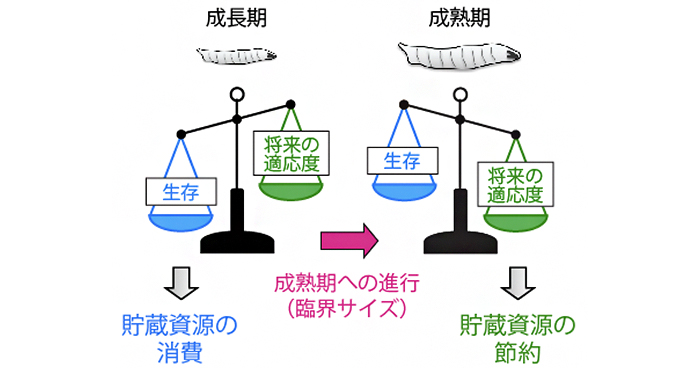
Lee SW, Morishita Y.
Possible roles of mechanical cell elimination intrinsic to growing tissues from the perspective of tissue growth efficiency and homeostasis.
Plos Computational Biology
13(7), e1005651 (2017)
doi: 10.1371/journal.pcbi.1005651
Morishita Y, Hironaka K, Lee SW, et al.
Reconstructing 3D deformation dynamics for curved epithelial sheet morphogenesis from positional data of sparsely-labeled cells.
Nature Communications
8, 15 (2017)
doi: 10.1038/s41467-017-00023-7
Morishita Y, Kuroiwa A, Suzuki T.
Quantitative analysis of tissue deformation dynamics reveals three characteristic growth modes and globally aligned anisotropic tissue deformation during chick limb development.
Development
142(9), 1672-1683 (2015)
doi: 10.1242/dev.109728
Morishita Y, Suzuki T.
Bayesian inference of whole-organ deformation dynamics from limited space-time point data.
Journal of Theoretical Biology
357, 74-85 (2014)
doi: 10.1016/j.jtbi.2014.04.027
Hironaka K, Morishita Y.
Encoding and decoding of positional information in morphogen-dependent patterning.
Current Opinion in Genetics and Development
22(6), 553-561 (2012)
doi: 10.1016/j.gde.2012.10.002
Morishita Y, Iwasa Y.
Coding Design of Positional Information for Robust Morphogenesis.
Biophysical Journal
101(10), 2324-2335 (2011)
doi: 10.1016/j.bpj.2011.09.048