
チームリーダー*
梅原 崇史
Ph.D.
エピジェネティクス制御研究チーム
[2023年3月 終了]
E-mail takashi.umehara[at]riken.jp
[at]を@に変えてください
*現:創薬タンパク質解析基盤ユニット 上級研究員
「エピジェネティクス(後成遺伝学)は、先天的な遺伝情報に「後付け」される生命情報に取り組む研究分野です。この「後付け」の主な実体は、クロマチンと呼ばれるゲノムDNA とヒストンタンパク質の複合体に対する化学修飾です。エピジェネティクスは癌やiPS 細胞、寿命の制御に関わることから、創薬や再生医療、アンチエイジングの分子標的として注目を集めています。私たちの研究室では、ヒトのエピジェネティクスの情報を精密に再現・検出・制御する技術の開発を通して、疾患やiPS 細胞、抗老化を望みどおりに操作することをめざします。
エピジェネティクスの再現
設計通りにアセチル化されたタンパク質の合成技術。この技術により、転写活性なクロマチンを試験管内で精密に再現することができる。
エピジェネティクスの検出
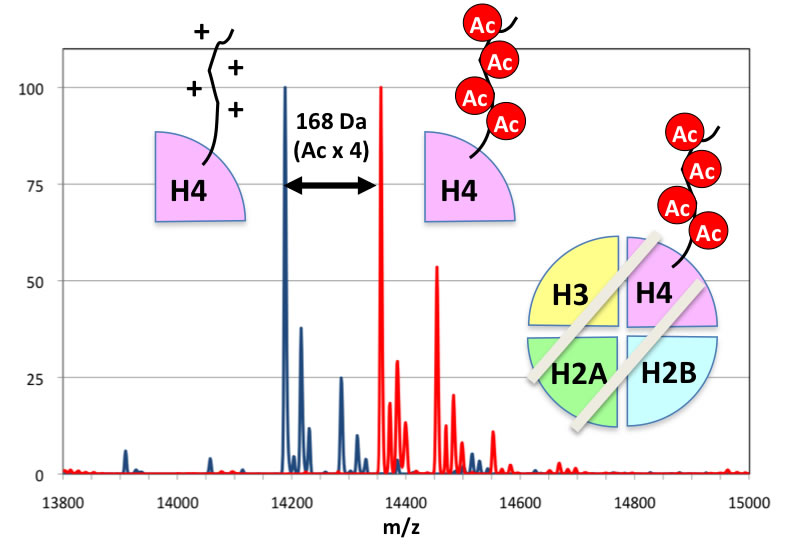
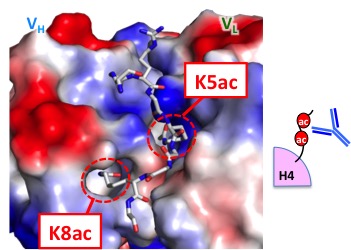
ヒストンH4 テイルの2 残基のアセチル化を検出する抗体の開発。この抗体を用いて、転写が超活性化したクロマチンを1 ヌクレオソーム分解能で検出することができる。
エピジェネティクスの制御
タンパク質の構造に基づいて開発したヒストン脱メチル化酵素阻害剤S2101。この化合物はエピジェネティクス制御試薬として市販されている。

主要論文
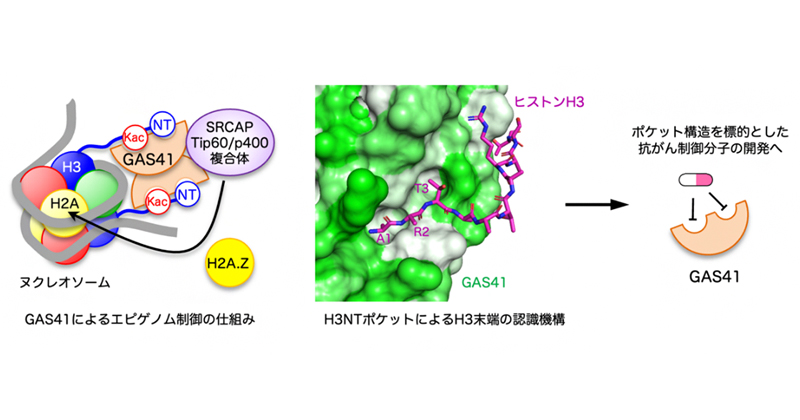
Kikuchi M, Takase S, Konuma T, et al.
GAS41 promotes H2A.Z deposition through recognition of the N terminus of histone H3 by the YEATS domain.
Proceedings of the National Academy of Sciences of the United States of America
120(43), e2304103120 (2023)
doi: 10.1073/pnas.2304103120
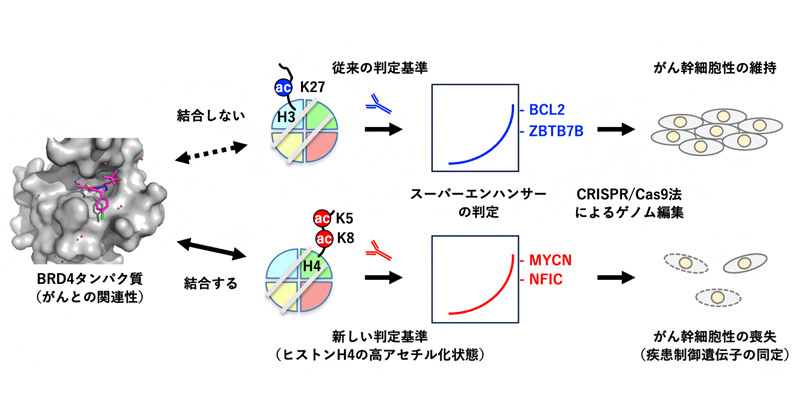
Das ND, Chang JC, Hon CC, et al.
Defining super-enhancers by highly ranked histone H4 multi-acetylation levels identifies transcription factors associated with glioblastoma stem-like properties.
BMC Genomics
24(1), 574 (2023)
doi: 10.1186/s12864-023-09659-w
Kikuchi M, Morita S, Wakamori M, et al.
Epigenetic mechanisms to propagate histone acetylation by p300/CBP.
Nature Communications
14(1), 4103 (2023)
doi: 10.1038/s41467-023-39735-4
Liu N, Konuma T, Sharma R, et al.
Histone H3 lysine 27 crotonylation mediates gene transcriptional repression in chromatin.
Molecular Cell
83(13), 2206-2221 (2023)
doi: 10.1016/j.molcel.2023.05.022
Noritsugu K, Suzuki T, Dodo K, et al.
Lysine long-chain fatty acylation regulates the TEAD transcription factor.
Cell Reports
42(4), 112388 (2023)
doi: 10.1016/j.celrep.2023.112388
Takase S, Hiroyama T, Shirai F, et al.
A specific G9a inhibitor unveils BGLT3 lncRNA as a universal mediator of chemically induced fetal globin gene expression.
Nature Communications
14(1), 23 (2023)
doi: 10.1038/s41467-022-35404-0
Niwa H, Watanabe C, Sato S, et al.
Structure–activity relationship and in silico evaluation of cis- and trans-PCPA-derived inhibitors of LSD1 and LSD2.
ACS Medicinal Chemistry Letters
13, 1485-1492 (2022)
doi: 10.1021/acsmedchemlett.2c00294
Kikuchi M, Morita S, Goto M, et al.
Elucidation of binding preferences of YEATS domains to site-specific acetylated nucleosome core particles.
Journal of Biological Chemistry
298, 102164 (2022)
doi: 10.1016/j.jbc.2022.102164
Sasaki K, Suzuki M, Sonoda T, et al.
Visualization of the dynamic interaction between nucleosomal histone H3K9 tri-methylation and HP1α chromodomain in living cells.
Cell Chemical Biology
29, 1153-1161 (2022)
doi: 10.1016/j.chembiol.2022.05.006
Koda Y, Sato S, Yamamoto H, et al.
Design and synthesis of tranylcypromine-derived LSD1 inhibitors with improved hERG and microsomal stability profiles.
ACS Medicinal Chemistry Letters
13, 848-854 (2022)
doi: 10.1021/acsmedchemlett.2c00120
Kitagawa H, Kikuchi M, Sato S, et al.
Structure-based identification of potent lysine-specific demethylase 1 inhibitor peptides and temporary cyclization to enhance proteolytic stability and cell growth-inhibitory activity.
Journal of Medicinal Chemistry
64, 3707-3719 (2021)
doi: 10.1021/acs.jmedchem.0c01371
Wakamori M, Okabe K, Ura K, et al.
Quantification of the effect of site-specific histone acetylation on chromatin transcription rate.
Nucleic Acids Research
48(22), 12648-12659 (2020)
doi: 10.1093/nar/gkaa1050
Wu HD, Kikuchi M, Dagliyan O, et al.
Rational design and implementation of a chemically inducible hetero-trimerization system.
Nature Methods
17, 928-936 (2020)
doi: 10.1038/s41592-020-0913-x
Furukawa A, Wakamori M, Arimura Y, et al.
Acetylated histone H4 tail enhances histone H3 tail acetylation by altering their mutual dynamics in the nucleosome.
Proceedings of the National Academy of Sciences USA
117, 19661-19663 (2020)
doi: 10.1073/pnas.2010506117
Saito S, Kikuchi J, Koyama D, et al.
Eradication of central nervous system leukemia of T-cell origin with a brain-permeable LSD1 inhibitor.
Clinical Cancer Research
25, 1601-1611 (2019)
doi: 10.1158/1078-0432.CCR-18-0919
Handoko L, Kaczkowski B, Hon CC, et al.
JQ1 affects BRD2-dependent and independent transcription regulation without disrupting H4-hyperacetylated chromatin states.
Epigenetics
13, 410-431 (2018)
doi: 10.1080/15592294.2018.1469891
ニュース
2023年10月17日 研究成果
エピゲノム修飾の位置を端から数える仕組み
2023年10月3日 研究成果
遺伝子発現を活性化するスーパーエンハンサーの再定義
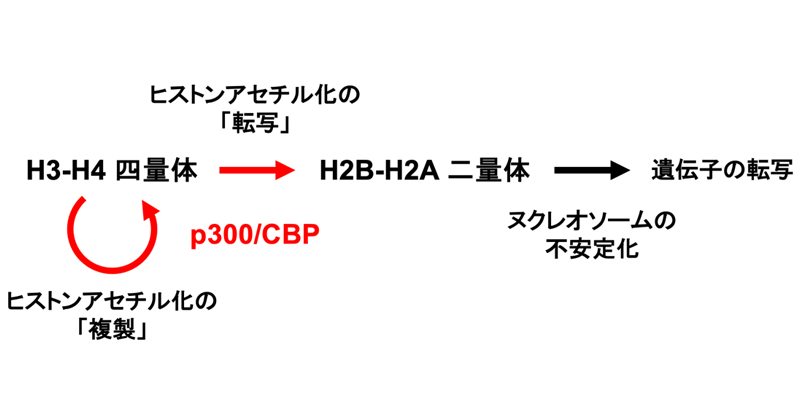
2023年7月17日 研究成果
遺伝子発現を制御するエピゲノムの複製と転写

2023年6月14日 研究成果
ヒストンH3タンパク質のクロトニル化修飾により大腸がん細胞が増幅する仕組みを解明
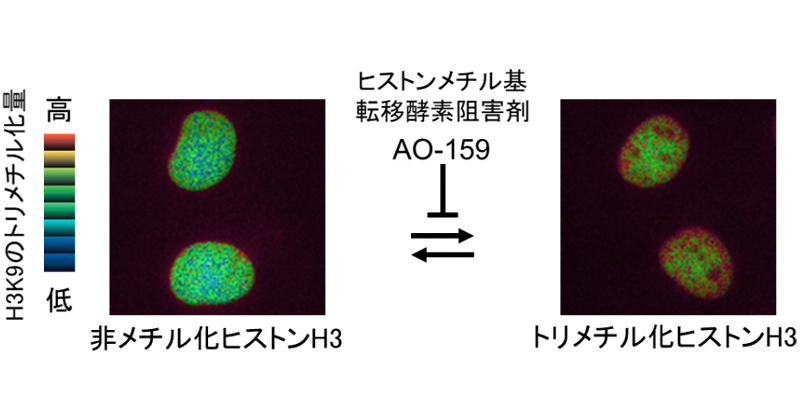
2022年6月21日 研究成果
生細胞内のヒストンメチル化を追う
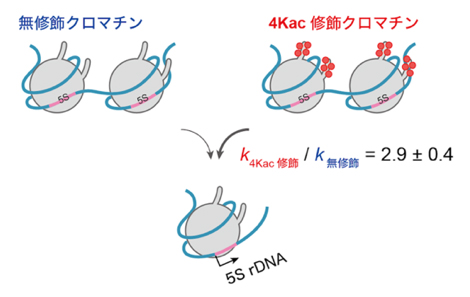
2020年11月26日 研究成果
エピゲノムの制御を受けた転写の方程式
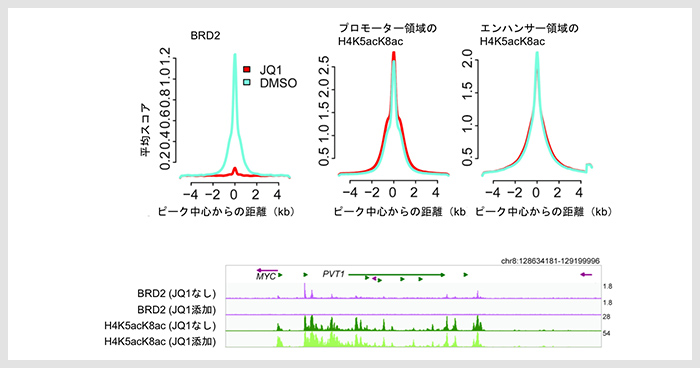
2018年8月20日 研究成果
がんエピゲノムのヒストンの高アセチル化は頑強だった
2018年8月8日 研究成果
「がんエピゲノム」を検出する新手法





