
タンパク質/ペプチドを自在に合成する技術基盤を構築
生命を理解するためには、細胞の内外において様々な生命機能の担い手として活躍するタンパク質の機能・構造への理解が不可欠です。当ユニットではタンパク質生合成を個々の因子から再構成した無細胞タンパク質合成システム、PUREシステムをベースに、タンパク質を自在に合成する技術基盤を構築し、タンパク質の機能制御・タンパク質の測定技術・分子モデリング技術などの進展に寄与することを目指しています。また、PUREシステムの構成要素をPUREシステムによって合成することによる無細胞タンパク質合成システムの自己複製モデルの構築を行うことによって、合成生物学・構成生物学が目指す生命システムの設計・制御のための技術基盤の構築に貢献することを目指しています。
研究テーマ
- 細胞内におけるタンパク質合成機構の解析
- PUREシステムを用いた有用タンパク質の高効率な生産手段の確立
- PUREシステムを用いたプロテオミクスツールの開発
- 無細胞タンパク質合成システムを用いた自己複製モデルの構築
主要論文
Okubo-Kurihara E, Ali A, Hiramoto M, et al.
Tracking metabolites at single-cell resolution reveals metabolic dynamics during plant mitosis.
Plant Physiology
189(2), 459-464 (2022)
doi: 10.1093/plphys/kiac093
Hibi K, Amikura K, Sugiura N, et al.
Reconstituted cell-free protein synthesis using in vitro transcribed tRNAs.
Communications Biology
3, 350 (2020)
doi: 10.1038/s42003-020-1074-2
Susaki EA, Shimizu C, Kuno A, et al.
Versatile whole-organ/body staining and imaging based on electrolyte-gel properties of biological tissues.
Nature Communications
11, 1982 (2020)
doi: 10.1038/s41467-020-15906-5
Shimojo M, Amikura K, Masuda K, et al.
In vitro reconstitution of functional small ribosomal subunit assembly for comprehensive analysis of ribosomal elements in E. coli.
Communications Biology
3, 142 (2020)
doi: 10.1038/s42003-020-0874-8
Narumi R, Masuda K, Tomonaga T, et al.
Cell-free synthesis of stable isotope-labeled internal standards for targeted quantitative proteomics.
Synthetic and Systems Biotechnology
3, 97-104 (2018)
doi: 10.1016/j.synbio.2018.02.004
Matsuura T, Tanimura N, Hosoda K, et al.
Reaction dynamics analysis of a reconstituted Escherichia coli protein translation system by computational modeling.
Proceedings of the National Academy of Sciences of the United States of America
114(8), E1336-E1344 (2017)
doi: 10.1073/pnas.1615351114
Narumi R, Shimizu Y, Ukai-Tadenuma M, et al.
Mass spectrometry-based absolute quantification reveals rhythmic variation of mouse circadian clock proteins.
Proceedings of the National Academy of Sciences of the United States of America
113(24), E3461-E3467 (2016)
doi: 10.1073/pnas.1603799113
Sunagawa GA, Sumiyama K, Ukai-Tadenuma M, et al.
Mammalian Reverse Genetics without Crossing Reveals Nr3a as a Short-Sleeper Gene.
Cell Reports
14(3), 662-677 (2016)
doi: 10.1016/j.celrep.2015.12.052
Tanaka Y, Shimizu Y.
Integration of a Reconstituted Cell-free Protein-synthesis System on a Glass Microchip.
Analytical Sciences
31(2), 67-71 (2015)
doi: 10.2116/analsci.31.67
Susaki EA, Tainaka K, Perrin D, et al.
Whole-Brain Imaging with Single-Cell Resolution Using Chemical Cocktails and Computational Analysis.
Cell
157(3), 726-739 (2014)
doi: 10.1016/j.cell.2014.03.042
Shimizu Y.
Biochemical aspects of bacterial strategies for handling the incomplete translation processes.
Frontiers in Microbiology
5, 1 (2014)
doi: 10.3389/fmicb.2014.00170
Shimizu Y, Kuruma Y, Kanamori T, Ueda T.
The PURE System for Protein Production.
Methods in Molecular Biology
1118, 275-284 (2014)
doi: 10.1007/978-1-62703-782-2_19
Shimizu Y.
ArfA Recruits RF2 into Stalled Ribosomes.
Journal of Molecular Biology
423(4), 624-631 (2012)
doi: 10.1016/j.jmb.2012.08.007
Kihira K, Shimizu Y, Shomura Y, et al.
Crystal structure analysis of the translation factor RF3 (release factor 3).
FEBS Letters
586(20), 3705-3709 (2012)
doi: 10.1016/j.febslet.2012.08.029
メンバー
清水 義宏
チームディレクター
益田 恵子
研究員
原田 頌子
研究員
西川 将太
研究員
中川 れい子
技師
佐藤 文
テクニカルスタッフⅠ
萬代 有美
テクニカルスタッフⅠ
坪井 節子
テクニカルスタッフⅠ
今里 亜貴子
研究パートタイマーⅡ
LAI Weng Yu
国際プログラム・アソシエイト
CHANG Tyng-Syuan
国際プログラム・アソシエイト
宮島 温人
理研スチューデント・リサーチャーM
長友 陽子
アシスタント
ニュース

2025年11月27日 研究成果
極小微生物のタンパク質生産装置は常識外れな組み立て方

2025年8月27日 研究成果
21種類の転移RNAの試験管内同時全合成を達成
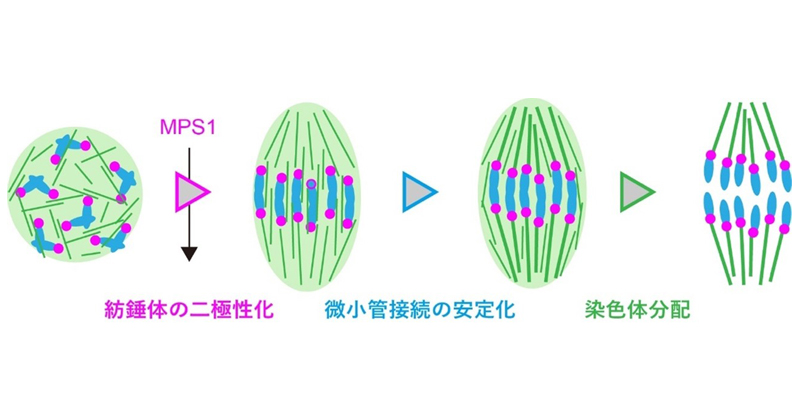
2025年6月4日 研究成果
卵子へ染色体を正しく分配する手順

2025年4月4日 研究成果
20種類の翻訳因子の持続的な再生産を達成

2024年9月10日 BDRニュース
BDRの研究ネホリハホリ
いろいろ計れる質量分析計

2024年6月10日 研究成果
複数のマイクロRNAを同時検出する「RNA液滴コンピュータ」の開発に成功

2022年6月14日 研究成果
生命現象を赤色光でコントロールする技術を開発

2022年4月15日 BDRニュース
文部科学大臣表彰の受賞について
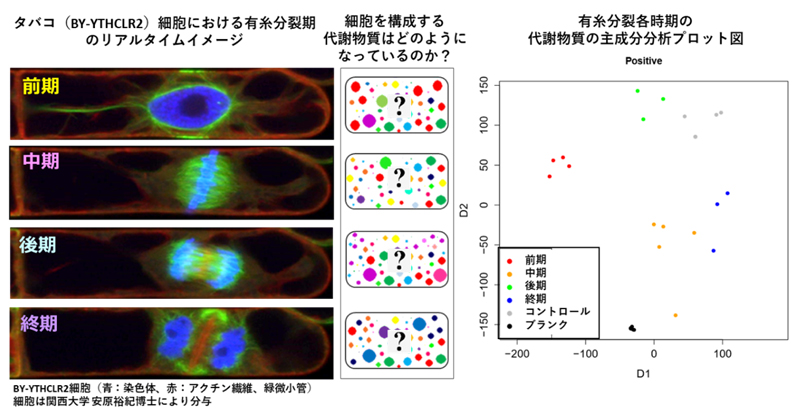
2022年3月18日 研究成果
植物の細胞分裂期の代謝物質を解明
2021年3月18日 研究成果
遺伝暗号を人工的につくる

2020年7月7日 研究成果
硫黄原子を導入した人工mRNAで高効率たんぱく質合成
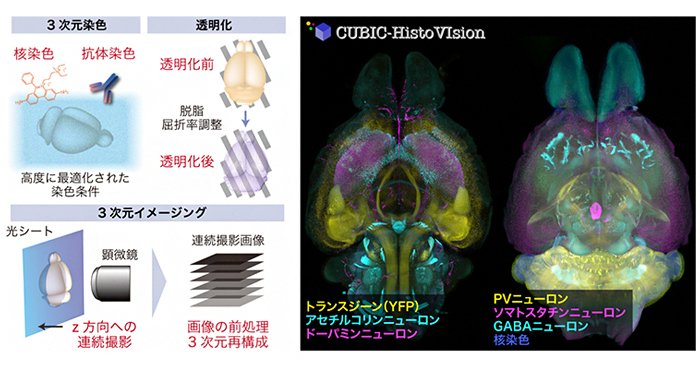
2020年4月27日 研究成果
3次元組織学による全臓器・全身の観察技術を確立
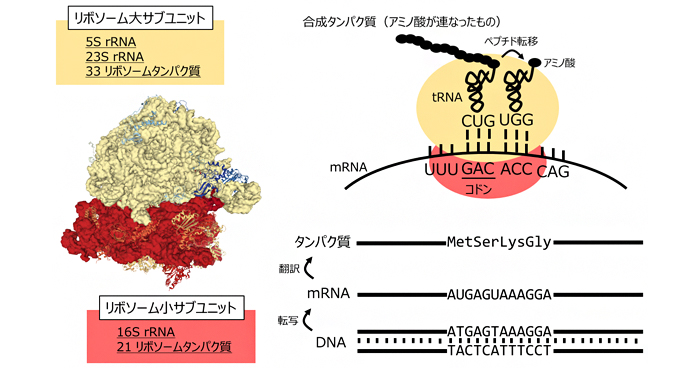
2020年3月25日 研究成果
リボソームを試験管内で自由に再構成
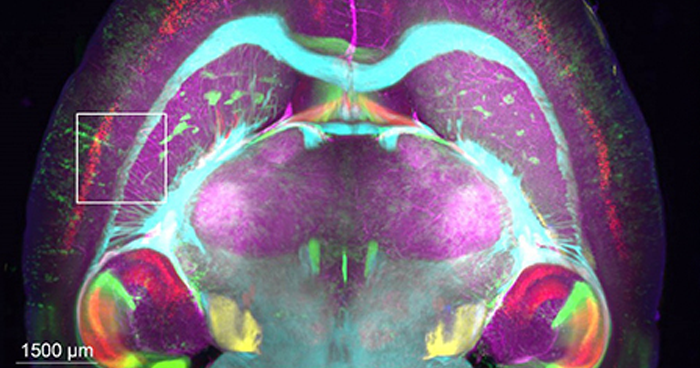
2018年8月22日 研究成果
水溶性化合物による組織透明化の化学






