チームディレクター
関根 俊一
Ph.D.
転写制御構造生物学研究チーム
[2025年4月よりBDRから生命医科学研究センター(IMS)に所属が変わりました]
拠点横浜
E-mail shunichi.sekine@riken.jp
生命活動は、生体を構成するタンパク質や核酸といった分子が集合したり、協調して働いたりすることで支えられています。その姿を可視化し、働く仕組みを明らかにするために、当チームでは、巨大な生体分子複合体の調製法を確立し、クライオ電子顕微鏡やSPring-8/SACLA の放射光を駆使して、それらの立体構造の解明を行なっています。特に、RNA ポリメラーゼと転写関連因子の複合体のような巨大複合体を解析し、転写やそれに関連する重要な現象の背後にある分子メカニズムを高分解能で解き明かします。
また、ウイルスや細菌が持つ複製・転写系のタンパク質は主要な創薬のターゲットのひとつであり、それらの立体構造解析により、創薬研究の基礎となる構造基盤を提供します。
研究テーマ
- RNAの転写およびそれに関連した生命現象の分子メカニズムの解明
- 超分子複合体の調製・解析技術の高度化
- ウイルスや細菌の複製・転写機構の解明と創薬への応用
主要論文
Naganuma M, Kujirai T, Ehara H, et al.
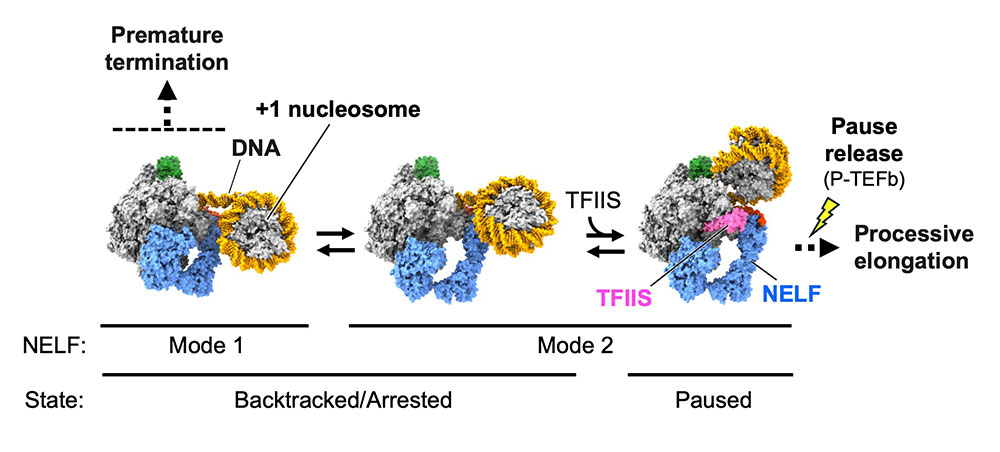
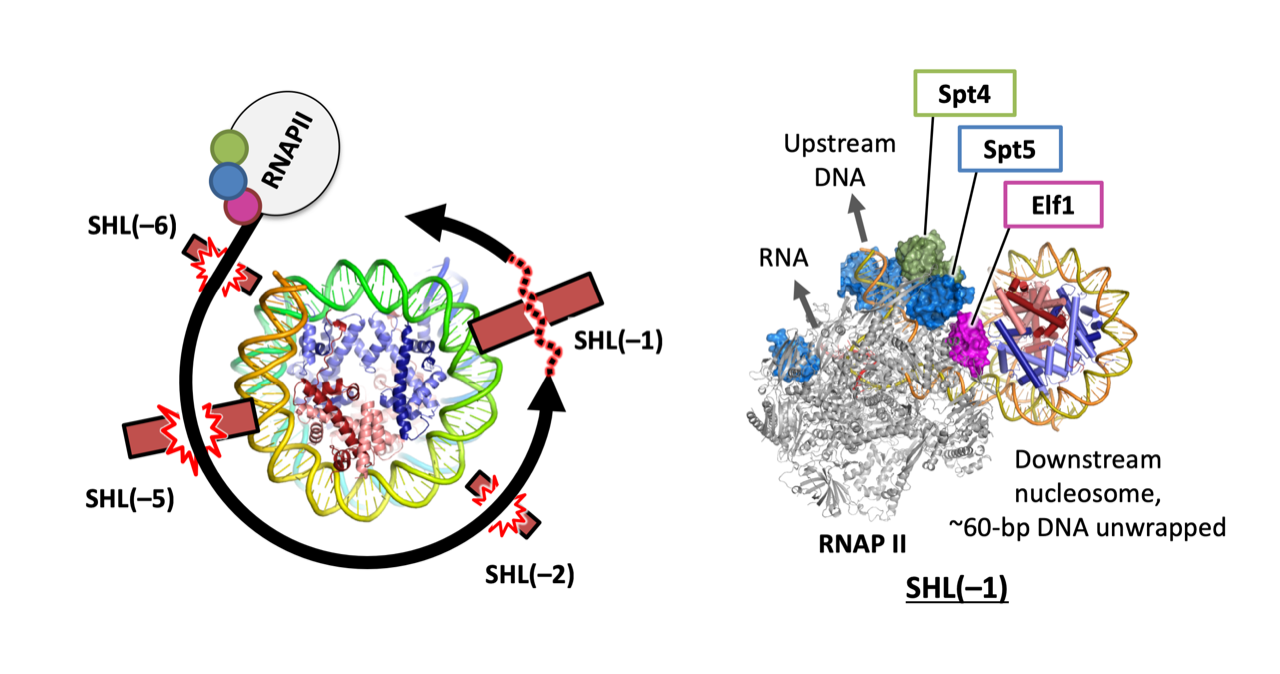
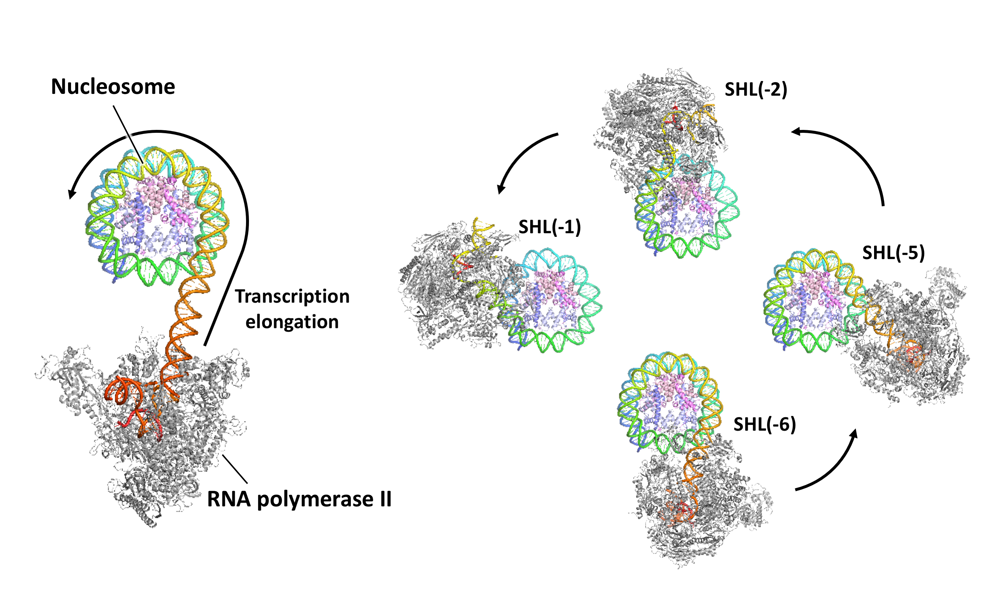
Structural insights into promoter-proximal pausing of RNA polymerase II at +1 nucleosome.
Science Advances
11(10), eadu0577 (2025)
doi: 10.1126/sciadv.adu0577
Yanagisawa T, Murayama Y, Ehara H, et al.
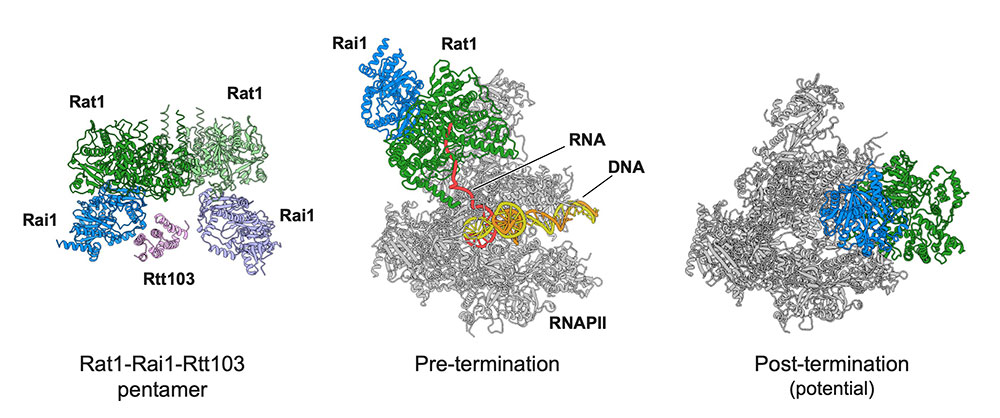
Structural basis of eukaryotic transcription termination by the Rat1 exonuclease complex
Nature Communications
15, 7854 (2024)
doi: 10.1038/s41467-024-52157-0
Akatsu M, Ehara H, Kujirai T, et al.
Cryo-EM structures of RNA polymerase II-nucleosome complexes rewrapping transcribed DNA
J Biol Chem
299(12), 105477 (2023)
doi: 10.1016/j.jbc.2023.105477
Sekine S, Ehara H, Kujirai T, et al.
Structural perspectives on transcription in chromatin
Trends in Cell Biology
(2023)
doi: 10.1016/j.tcb.2023.07.011
Osawa T, Aoki M, Ehara H, et al.
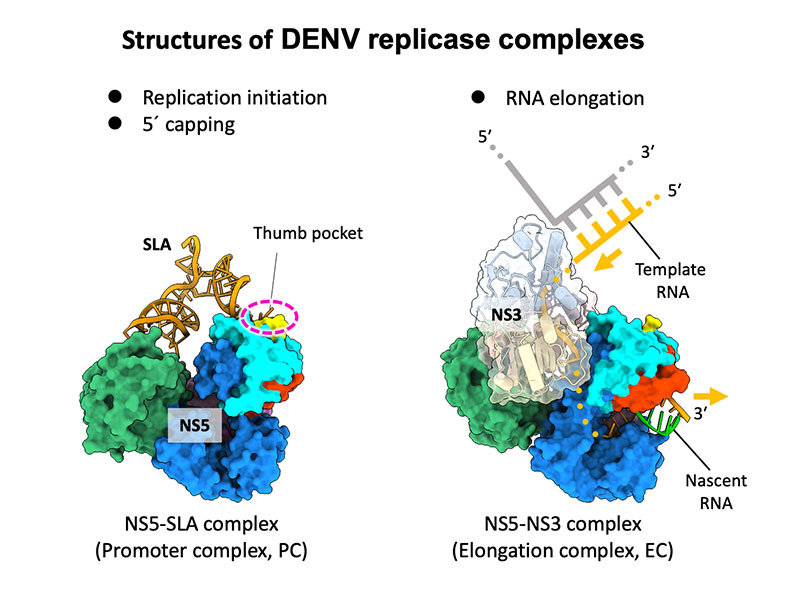
Structures of dengue virus RNA replicase complexes
Molecular Cell
(2023)
doi: 10.1016/j.molcel.2023.06.023
Murayama Y, Ehara H, Aoki M, et al.
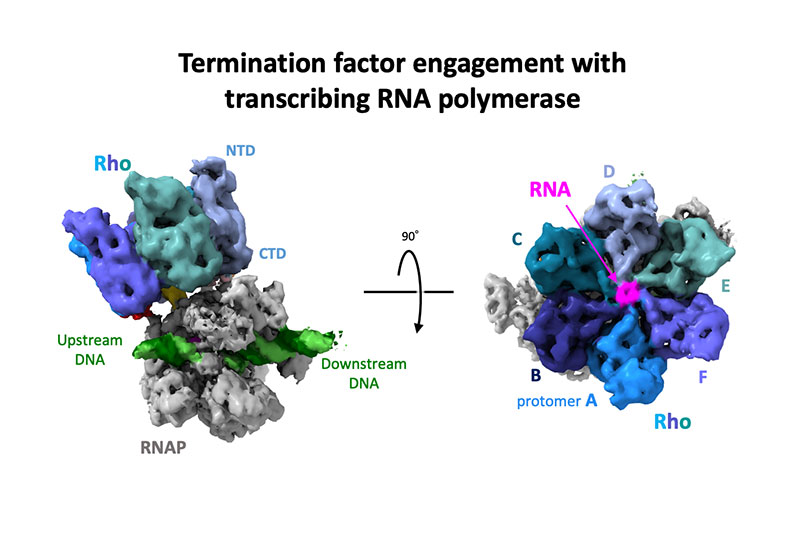
Structural basis of the transcription termination factor Rho engagement with transcribing RNA polymerase from Thermus thermophilus
Science Advances
9(6), eade7093 (2023)
doi: 10.1126/sciadv.ade7093
Hirano R, Ehara H, Kujirai T, et al.
Structural basis of RNA polymerase II transcription on the chromatosome containing linker histone H1
Nature Communications
13, 7287 (2022)
doi: 10.1038/s41467-022-35003-z
Ehara H, Kujirai T, et al.
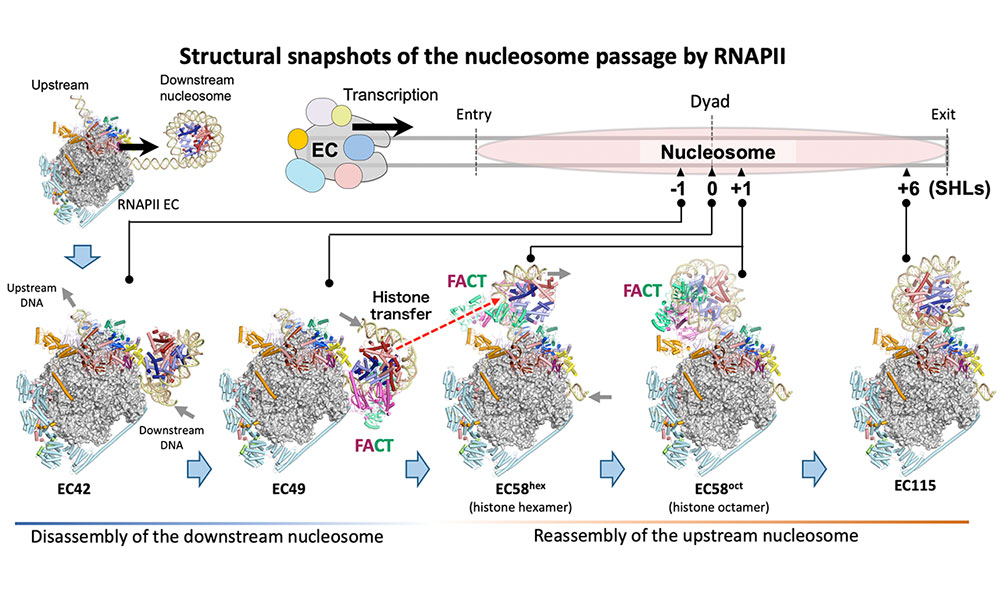
Structural basis of nucleosome disassembly and reassembly by RNAPII elongation complex with FACT
Science
377(6611), abp9466 (2022)
doi: 10.1126/science.abp9466
Ehara H, Kujirai T, et al.
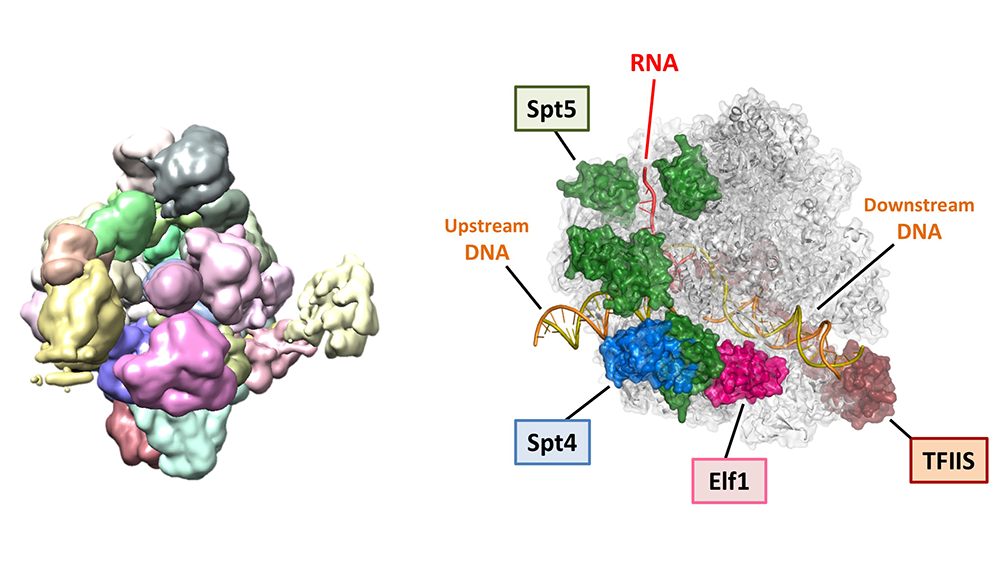
Structural insight into nucleosome transcription by RNA polymerase II with elongation factors.
Science
363(6428), 744-747 (2019)
doi: 10.1126/science.aav8912
Shimizu H, Saito A, Mikuni J, et al.
Discovery of a small molecule inhibitor targeting dengue virus NS5 RNA-dependent RNA polymerase.
Plos Neglected Tropical Diseases
13(11), e0007894 (2019)
doi: 10.1371/journal.pntd.0007894
Kujirai T, Ehara H, et al.
Structural basis of the nucleosome transition during RNA polymerase II passage.
Science
362(6414), 595-598 (2018)
doi: 10.1126/science.aau9904
Ooi WY, Murayama Y, Mekler V, et al.
A Thermus phage protein inhibits host RNA polymerase by preventing template DNA strand loading during open promoter complex formation.
Nucleic Acids Research
46(1), 431-441 (2017)
doi: 10.1093/nar/gkx1162
Ehara H, Yokoyama T, Shigematsu H, et al.
Structure of the complete elongation complex of RNA polymerase II with basal factors.
Science
357(6354), 921-924 (2017)
doi: 10.1126/science.aan8552
Sekine S, Murayama Y, Svetlov V, et al.
The ratcheted and ratchetable structural states of RNA polymerase underlie multiple transcriptional functions.
Molecular Cell
57(3), 408-421 (2015)
doi: 10.1016/j.molcel.2014.12.014
メンバー

チームディレクター関根 俊一
- shunichi.sekine@riken.jp
- CV
ニュース

2025年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞、理研桜舞賞の授与について
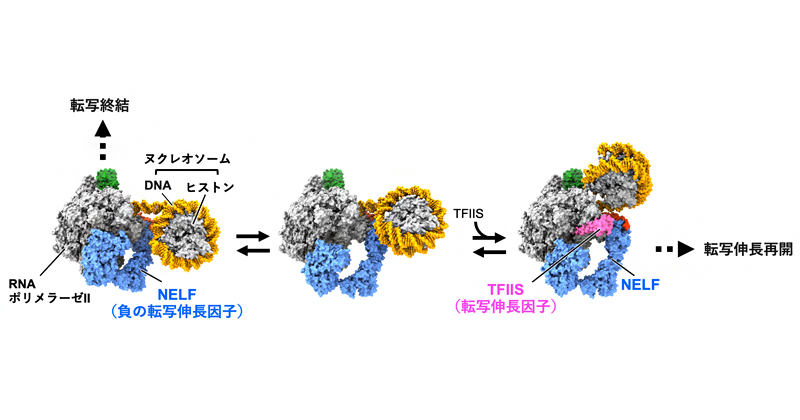
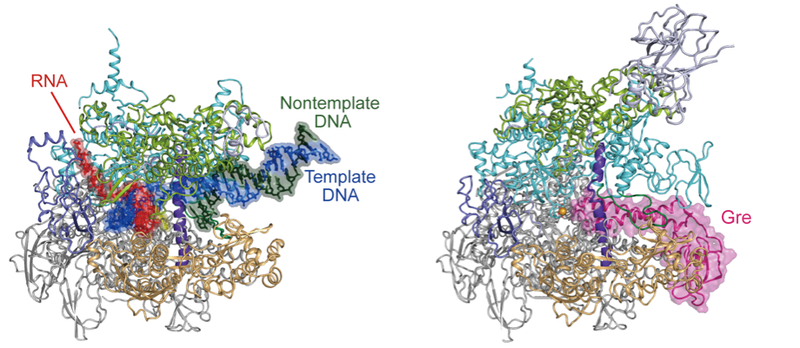
2025年3月6日 研究成果
RNAポリメラーゼに一時停止を守らせる仕組み
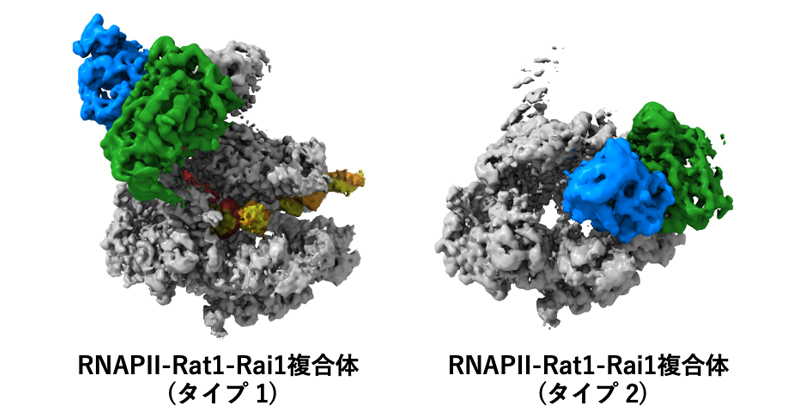
2024年9月20日 研究成果
真核生物での遺伝子の読み取りが終わる瞬間を捉える

2024年4月22日 BDRニュース
文部科学大臣表彰の受賞について

2024年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について
2023年9月21日 BDRニュース
研究最前線
転写の瞬間を撮影して、生物学上の長年の謎を解決!
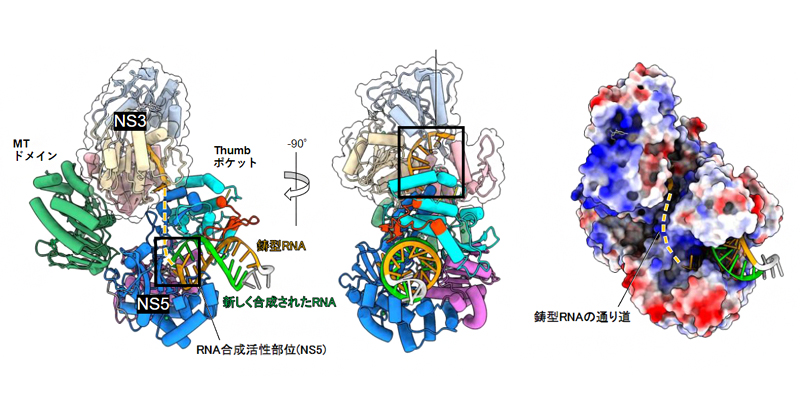
2023年7月21日 研究成果
デングウイルスのRNA複製酵素の立体構造を解明

2023年3月23日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について
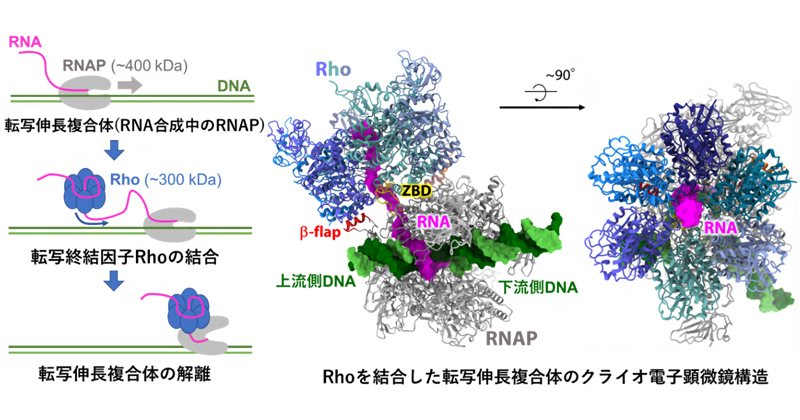
2023年2月9日 研究成果
遺伝子の読み取りを終わらせるメカニズム

2022年11月30日 研究成果
染色体の中で折りたたまれたDNAから遺伝情報を読み取る仕組みを解明!
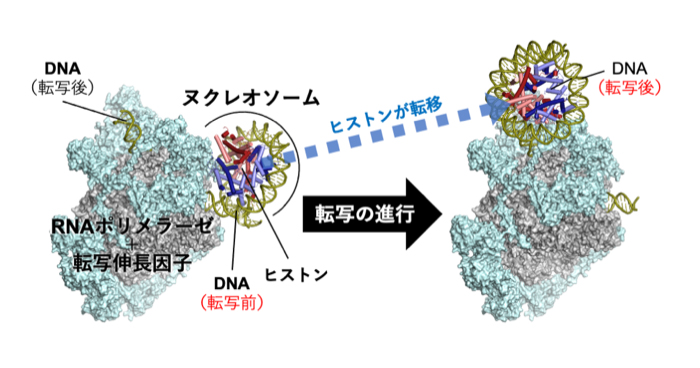
2022年8月19日 研究成果
遺伝子の発現とクロマチン構造の維持を両立させる仕組み

2022年4月15日 BDRニュース
文部科学大臣表彰の受賞について
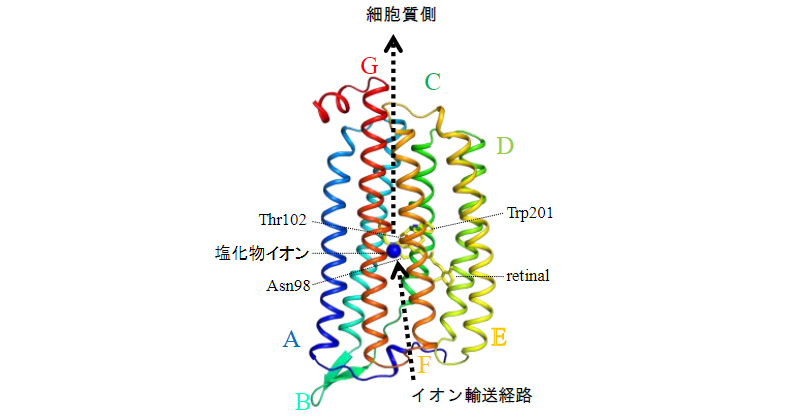
2022年2月28日 研究成果
光でイオンを輸送する膜タンパク質の巧妙な仕組み

2021年11月1日 BDRニュース
BDRの研究ネホリハホリ
巨人の肩に乗る-クライオ電顕の躍進

2019年6月12日 BDRニュース
2018年度「理研栄峰賞」および2018年度「理研梅峰賞」の受賞者が決まりました
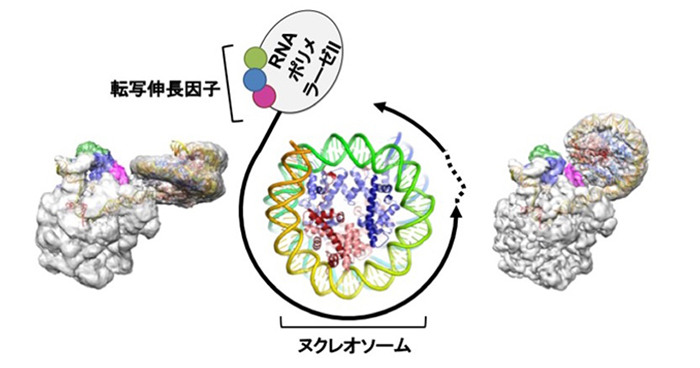
2019年2月8日 研究成果
コンパクトなDNAをスムーズに転写する仕組み
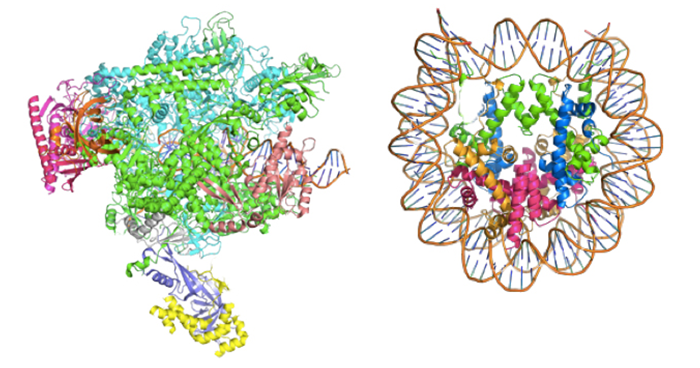
2018年10月5日 研究成果
真核生物での遺伝子読み取りの仕組みを解明