
チームリーダー
上田 昌宏
Ph.D.
細胞シグナル動態研究チーム
[2025年3月 終了]
E-mail masahiroueda@riken.jp
本研究グループは、細胞のシグナル伝達機能が生体分子の反応ネットワークのダイナミクスによって自発的に生み出される仕組み(構築原理)と、その情報処理システムが確率的なゆらぎの影響を強く受けながらも機能する仕組み(演算原理)に興味を持っています。こうした生体分子による確率的な演算の典型例として、我々は細胞性粘菌における走化性シグナル伝達系とヒト細胞における成長因子のシグナル伝達系に焦点をあて研究を進めています。1分子計測技術などを含む最先端のイメージング技術と理論・高性能計算機を用いた数理モデル構築を組み合わせることにより、細胞のシグナル伝達を担うシステムの構築原理と演算原理の理解を目指した新しい研究手法の開発を行ないます。
研究テーマ
- 細胞における自発的シグナル形成
- 確率的シグナル伝達とプロセッシング
- 細胞における自動1分子イメージング解析
- 生物システムの階層構造における「構造化されたランダムネス」
主要論文
Miyagawa T, Koteishi H, Kamimura Y, et al.
Structural basis of Gip1 for cytosolic sequestration of G protein in wide-range chemotaxis.
Nature Communications
9, 4635 (2018)
doi: 10.1038/s41467-018-07035-x
Matsuoka S, Ueda M.
Mutual inhibition between PTEN and PIP3 generates bistability for polarity in motile cells.
Nature communications
9(1), 4481 (2018)
doi: 10.1038/s41467-018-06856-0
Yasui M, Hiroshima M, Kozuka J, et al.
Automated single-molecule imaging in living cells.
Nature Communications
9, 3061 (2018)
doi: 10.1038/s41467-018-05524-7
Yanagawa M, Hiroshima M, Togashi Y, et al.
Single-molecule diffusion-based estimation of ligand effects on G protein-coupled receptors.
Science signaling
11(548), eaao1917 (2018)
doi: 10.1126/scisignal.aao1917
Kamimura Y, Miyanaga Y, Ueda M.
Heterotrimeric G-protein shuttling via Gip1 extends the dynamic range of eukaryotic chemotaxis.
Proceedings of the National Academy of Sciences of the United States of America
113(16), 4356-4361 (2016)
doi: 10.1073/pnas.1516767113
Komatsuzaki A, Ohyanagi T, Tsukasaki Y, et al.
Compact Halo-ligand-conjugated quantum dots for multicolored single-molecule imaging of overcrowding GPCR proteins on cell membranes.
Small
11(12), 1396-1401 (2015)
doi: 10.1002/smll.201402508
Yasui M, Matsuoka S, Ueda M.
PTEN Hopping on the Cell Membrane Is Regulated via a Positively-Charged C2 Domain.
Plos Computational Biology
10(9), e1003817 (2014)
doi: 10.1371/journal.pcbi.1003817
Cai H, Katoh-Kurasawa M, Muramoto T, et al.
Nucleocytoplasmic Shuttling of a GATA Transcription Factor Functions as a Development Timer.
Science
343(6177), 1329-+ (2014)
doi: 10.1126/science.1249531
Nishikawa M, Horning M, Ueda M, Shibata T.
Excitable Signal Transduction Induces Both Spontaneous and Directional Cell Asymmetries in the Phosphatidylinositol Lipid Signaling System for Eukaryotic Chemotaxis.
Biophysical Journal
106(3), 723-734 (2014)
doi: 10.1016/j.bpj.2013.12.023
Okamoto M, Namba T, Shinoda T, et al.
TAG-1-assisted progenitor elongation streamlines nuclear migration to optimize subapical crowding.
Nature Neuroscience
16(11), 1556-1566 (2013)
doi: 10.1038/nn.3525
Matsuoka S, Shibata T, Ueda M.
Asymmetric PTEN Distribution Regulated by Spatial Heterogeneity in Membrane-Binding State Transitions.
Plos Computational Biology
9(1), e1002862 (2013)
doi: 10.1371/journal.pcbi.1002862
Nishimura SI, Ueda M, Sasai M.
Non-Brownian dynamics and strategy of amoeboid cell locomotion.
Physical Review E
85(4), 041909 (2012)
doi: 10.1103/PhysRevE.85.041909
Tsujioka M, Yumura S, Inouye K, et al.
Talin couples the actomyosin cortex to the plasma membrane during rear retraction and cytokinesis.
Proceedings of the National Academy of Sciences of the United States of America
109(32), 12992-12997 (2012)
doi: 10.1073/pnas.1208296109
Arai Y, Shibata T, Matsuoka S, et al.
Self-organization of the phosphatidylinositol lipids signaling system for random cell migration.
Proceedings of the National Academy of Sciences of the United States of America
107(27), 12399-12404 (2010)
doi: 10.1073/pnas.0908278107
ニュース

2025年1月17日 研究成果
細胞の自発的なアメーバ運動を駆動するRas活性化タンパク質を発見

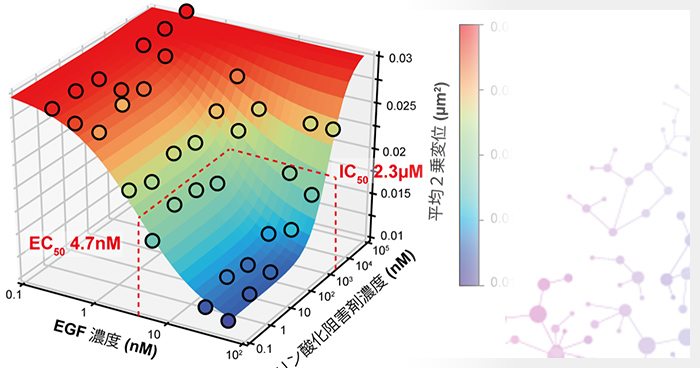
2024年11月1日 研究成果
大規模細胞内1分子イメージングによる新規薬剤スクリーニングを実現
2024年1月10日 研究成果
命名“Dynamic partitioning”
膜タンパク質が局在化するしくみを解明

2022年1月21日 研究成果
シグナル伝達による多様な細胞応答の起源

2020年1月20日 BDRニュース
研究者にズームイン
AIと細胞

2019年6月12日 BDRニュース
2018年度「理研栄峰賞」および2018年度「理研梅峰賞」の受賞者が決まりました
2019年1月31日 研究成果
細胞の中で機能している分子の観察を完全自動化する

2018年11月22日 研究成果
シグナル伝達に関わる三量体Gタンパク質が細胞膜を離れて細胞質にとどまる仕組みを原子レベルで解明
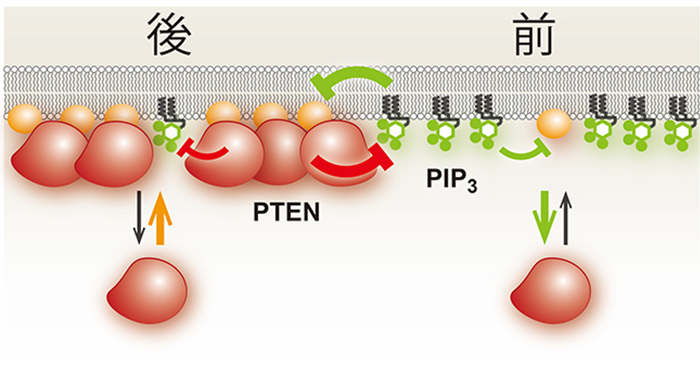
2018年10月30日 研究成果
運動する細胞の進行方向を決める仕組みを解明

2018年9月26日 研究成果
細胞内1分子自動観察システム「AiSIS」
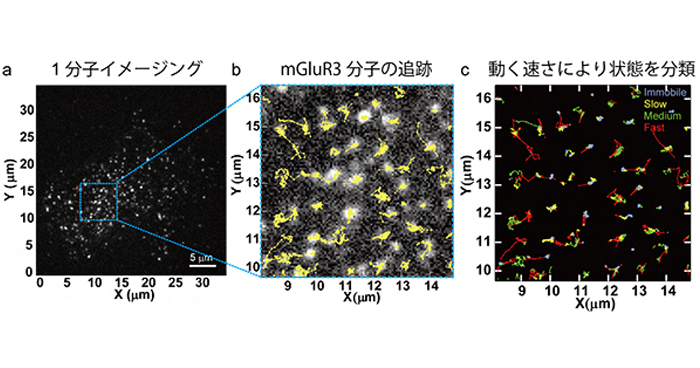
2018年9月19日 研究成果
細胞膜の受容体1分子の動きから薬効を評価





