
チームリーダー
上田 泰己
M.D., Ph.D.
合成生物学研究チーム
[2025年3月 終了]
E-mail uedah-tky@umin.ac.jp
生命科学研究は還元論的アプローチから、生命の部品を組み合わせて生命現象を再現・設計する構成論的アプローチへパラダイムシフトが起こりつつあります。合成生物学が新しい生命科学研究の領域として認識されつつある一方で、遺伝子やタンパク質などの生命の部品を調整・設計・制御し、生命を創るための技術は十分確立されていません。当グループでは、このような状況を鑑み、概日時計や睡眠現象をモデル系として生命システムの制御および設計の先見的な実現例を示すとともに、合成生物学の基盤テクノロジーを開発することを目指します。
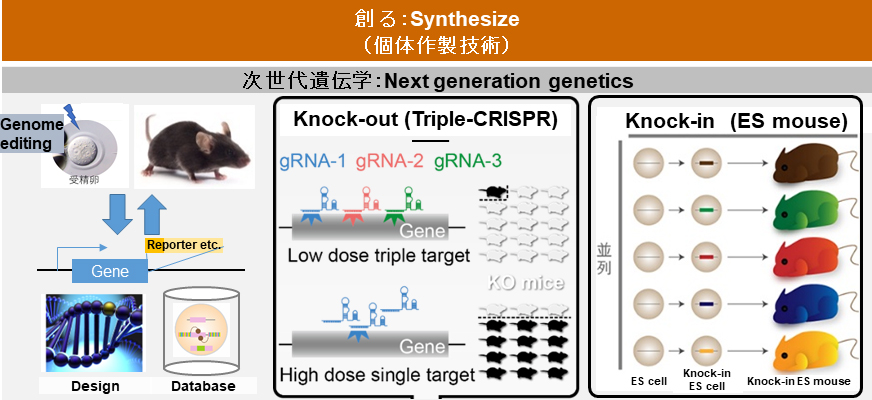

研究テーマ
- タンパク質動態の解析に基づいたタンパク質活性制御化合物および新規機能タンパク質の設計
- 概日時計をモデルとしたタンパク質複合体の設計
- 遺伝子ネットワーク改変技術の開発
- 生命機能のデザインと構築に向けた新規遺伝子合成システムの開発
- 1細胞レベルでのタンパク質ネットワークの構築および動態測定
主要論文
Susaki EA, Shimizu C, Kuno A, et al.
Versatile whole-organ/body staining and imaging based on electrolyte-gel properties of biological tissues.
Nature Communications
11, 1982 (2020)
doi: 10.1038/s41467-020-15906-5
Funano SI, Tone D, Ukai H, et al.
Rapid and easy-to-use ES cell manipulation device with a small groove near culturing wells.
BMC research notes
13(1), 453 (2020)
doi: 10.1186/s13104-020-05294-w
Ota N, Kanda GN, Moriguchi H, et al.
A Microfluidic Platform Based on Robust Gas and Liquid Exchange for Long-term Culturing of Explanted Tissues.
Analytical sciences : the international journal of the Japan Society for Analytical Chemistry
35(10), 1141-1147 (2019)
doi: 10.2116/analsci.19P099
Matsumoto K, Mitani TT, Horiguchi SA, et al.
Advanced CUBIC tissue clearing for whole-organ cell profiling.
Nature Protocols
14, 3506-3537 (2019)
doi: 10.1038/s41596-019-0240-9
Niwa Y, Kanda GN, Yamada RG, et al.
Muscarinic Acetylcholine Receptors Chrm1 and Chrm3 Are Essential for REM Sleep.
Cell reports
24(9), 2231-2247 (2018)
doi: 10.1016/j.celrep.2018.07.082
Tainaka K, Murakami TC, Susaki EA, et al.
Chemical Landscape for Tissue Clearing Based on Hydrophilic Reagents.
Cell reports
24(8), 2196-2210 (2018)
doi: 10.1016/j.celrep.2018.07.056
Ukai H, Kiyonari H, Ueda HR.
Production of knock-in mice in a single generation from embryonic stem cells.
Nature Protocol
12, 2513-2530 (2017)
doi: 10.1038/nprot.2017.110
Shinohara Y, Koyama YM, Ukai-Tadenuma M et al.
Temperature-Sensitive Substrate and Product Binding Underlie Temperature-Compensated Phosphorylation in the Clock.
Molecular Cell
67, 783-798 (2017)
doi: 10.1016/j.molcel.2017.08.009
Kubota SI, Takahashi K, et al.
Whole-Body Profiling of Cancer Metastasis with Single-Cell Resolution.
Cell Reports
20, 236-250 (2017)
doi: 10.1016/j.celrep.2017.06.010
Ode KL, Ukai H, Susaki EA, et al.
Knockout-rescue embryonic stem cell-derived mouse reveals circadian-period control by quality and quantity of CRY1.
Molecular Cell
65, 176-190 (2017)
doi: 10.1016/j.molcel.2016.11.022
Narumi R, Shimizu Y, et al.
Mass spectrometry-based absolute quantification reveals rhythmic variation of mouse circadian clock proteins.
Proceedings National Academy of Sciences of the United States of America
133, E3461-E3467 (2016)
doi: 10.1073/pnas.1603799113
Tatsuki F, Sunagawa GA, Shi S, et al.
Involvement of Ca2+-Dependent Hyperpolarization in Sleep Duration in Mammals.
Neuron
90(1), 70-85 (2016)
doi: 10.1016/j.neuron.2016.02.032
Sunagawa GA.
Mammalian Reverse Genetics without Crossing Reveals Nr3a as a Short-Sleeper Gene.
Cell Reports
14(3), 662-677 (2016)
doi: 10.1016/j.celrep.2015.12.052
Susaki EA, Tainaka K, Perrin D, et al.
Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging.
Nature Protocol
10(11), 1709-1727 (2015)
doi: 10.1038/nprot.2015.085
Tainaka K, Kubota SI, Suyama TQ, et al.
Whole-Body Imaging with Single-Cell Resolution by Tissue Decolorization.
Cell
159(4), 911-924 (2014)
doi: 10.1016/j.cell.2014.10.034
Susaki EA, Tainaka K, Perrin D, et al.
Whole-Brain Imaging with Single-Cell Resolution Using Chemical Cocktails and Computational Analysis.
Cell
157(3), 726-739 (2014)
doi: 10.1016/j.cell.2014.03.042
ニュース
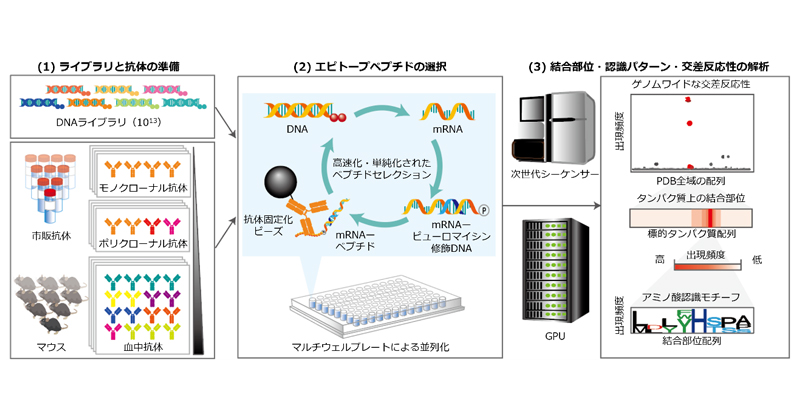
2025年2月6日 研究成果
単一アミノ酸分解能かつ網羅的なエピトープ解析法の開発

2024年12月3日 研究成果
マウス臓器の透明化と解析のプロトコルを確立

2024年11月7日 研究成果
哺乳類の睡眠・覚醒をリン酸化・脱リン酸化酵素群が制御

2024年9月3日 BDRニュース
起立性調節障害の子どもを対象とした「子ども睡眠検診」プロジェクトを開始

2024年7月22日 研究成果
徹夜後に長く深く眠る仕組みを解明

2024年3月18日 BDRニュース
「子ども睡眠健診」プロジェクトで見えてきた実態
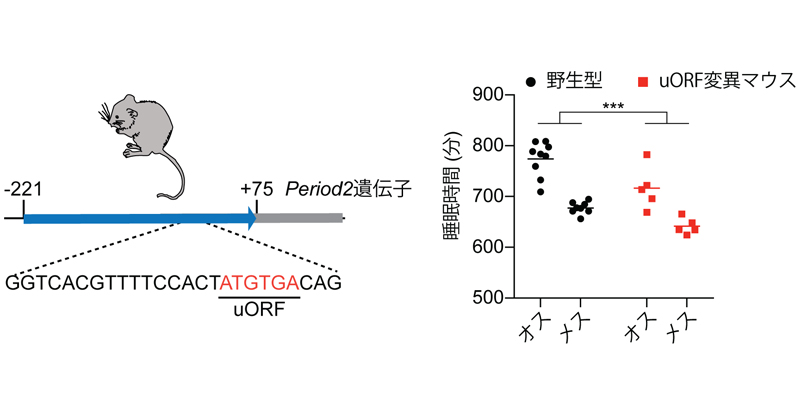
2023年10月25日 研究成果
睡眠制御における転写後プロセスの役割を解明

2023年9月4日 BDRニュース
「子ども睡眠健診」プロジェクト参加校(小・中・高)の第二次(2024年度前期)募集を開始

2022年10月5日 研究成果
睡眠に関わるたんぱく質リン酸化酵素の働きを解明

2022年9月12日 BDRニュース
日本全国の子ども(小中高生)を対象とした「子ども睡眠健診」プロジェクトを開始

2022年6月9日 BDRニュース
研究者にズームイン
夢見る夢への夢の研究
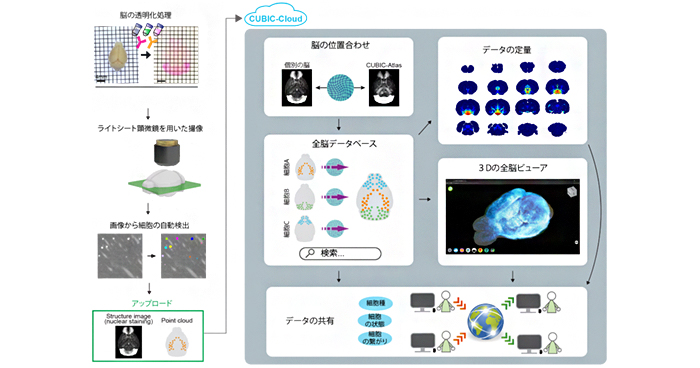
2021年6月22日 研究成果
脳の全細胞を解析するクラウドシステムの開発

2021年3月18日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について
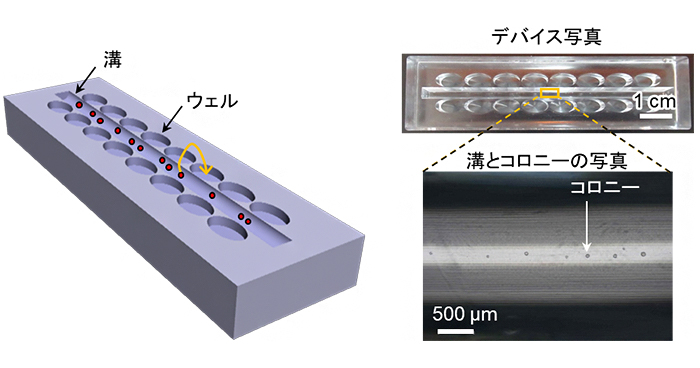
2020年10月5日 研究成果
ES細胞塊を簡単に単離できるデバイス

2020年9月23日 BDRニュース
「羽田みらいサイエンスギャラリー」がオープン
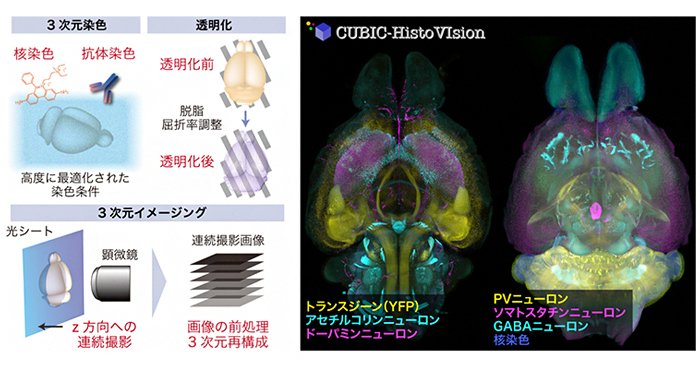
2020年4月27日 研究成果
3次元組織学による全臓器・全身の観察技術を確立

2019年12月13日 研究成果
臓器内の全細胞を調べる革新技術
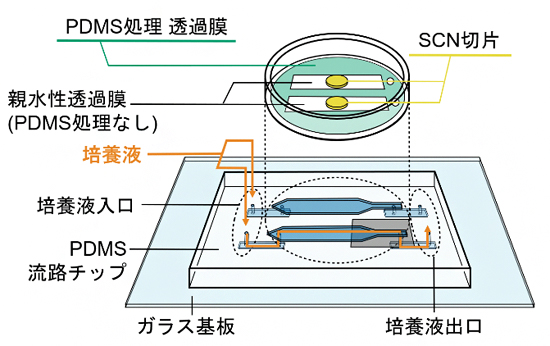
2019年10月10日 研究成果
マイクロ流体デバイスで生物組織を簡単に長期培養

2019年5月18日 BDRニュース
山田陸裕 上級研究員(合成生物学研究チーム)のエッセイが産経新聞の連載「科学の中身」に掲載されました
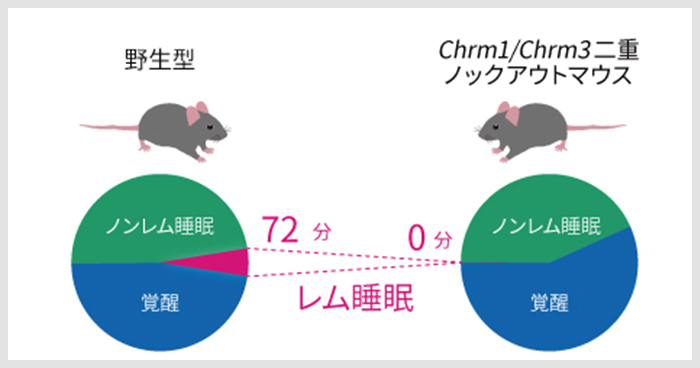
2018年10月16日 研究成果
レム睡眠を引き起こす2つの遺伝子
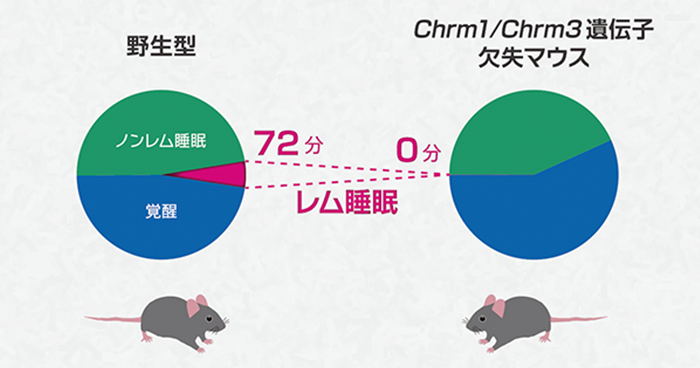
2018年8月29日 研究成果
レム睡眠に必須な遺伝子を発見
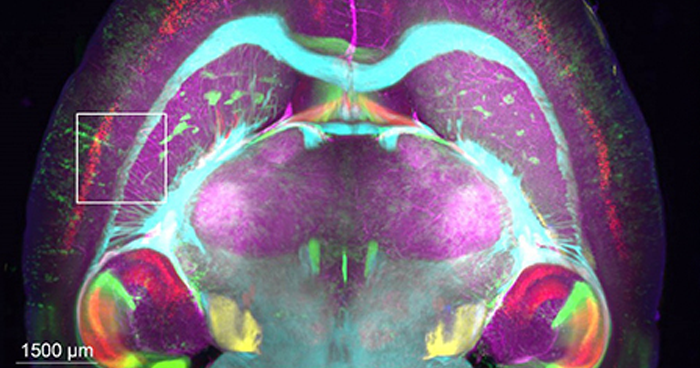
2018年8月22日 研究成果
水溶性化合物による組織透明化の化学

2018年7月5日 BDRニュース
理研ニュース7月号に上田泰己チームリーダー(合成生物学研究チーム)らの研究が紹介されました





