
多細胞生物の発生は時間的空間的に動的な過程です。受精卵と呼ばれる一つの細胞は細胞分裂を繰り返して様々な機能を持つ細胞を作り、それらが特定の位置に配置されることにより、複雑な構造を持つ器官や個体が作られます。このような時間的空間的に動的な過程のメカニズムを理解するためには、現象の定量化と数理モデル化、計算機シミュレーションを組み合わせた定量的計算科学的アプローチが有効です。当研究チームは、分子細胞生物学、生物物理学、ゲノム科学、計算科学、数理科学等の研究手法を統合的に用い、線虫 C. elegans胚やマウス胚、立体培養系等をモデル系として発生システムの数理モデルを構築し、多細胞生物の発生のメカニズムの解明を目指します。
研究テーマ
- 大量の定量動態情報を利用した発生のシステム解析
- 発生の数理モデルの構築
- 発生動態の定量化技術の開発
主要論文
Kyoda K, Itoga H, Yamagata Y, et al.
SSBD: an ecosystem for enhanced sharing and reuse of bioimaging data.
Nucleic Acids Research
53(D1), D1716-D1723 (2025)
doi: 10.1093/nar/gkae860
Shinkai S, Onami S, Miyaguchi T.
Generalized Langevin dynamics for single beads in linear elastic networks.
Physical Review. E
110(4), 044136 (2024)
doi: 10.1103/PhysRevE.110.044136
Nozaki T, Shinkai S, Ide S, et al.
Condensed but liquid-like domain organization of active chromatin regions in living human cells.
Science Advances
9(14), eadf1488 (2023)
doi: 10.1126/sciadv.adf1488
Hirata T, Tohsato Y, Itoga H, et al.
NeuroGT: A brain atlas of neurogenic tagging CreER drivers for birthdate-based classification and manipulation of mouse neurons.
Cell Reports Methods
1, 100012 (2021)
doi: 10.1016/j.crmeth.2021.100012
Swedlow JR, Kankaanpää P, Sarkans U, et al.
A global view of standards for open image data formats and repositories.
Nature Methods
18, 1440-1446 (2021)
doi: 10.1038/s41592-021-01113-7
Shinkai S, Nakagawa M, Sugawara T, et al.
PHi-C: deciphering Hi-C data into polymer dynamics.
NAR Genomics and Bioinformatics
2, lqaa020 (2020)
doi: 10.1093/nargab/lqaa020
Takayama J, Onami S.
The sperm TRP-3 channel mediates the onset of a Ca2+ wave in the fertilized C. elegans oocyte.
Cell Reports
15(3), 625-637 (2016)
doi: 10.1016/j.celrep.2016.03.040
Tohsato Y, Ho KHL, Kyoda K, Onami S.
SSBD: a database of quantitative data of spatiotemporal dynamics of biological phenomena.
Bioinformatics
32(22), 3471-3479 (2016)
doi: 10.1093/bioinformatics/btw417
Kyoda K, Adachi E, Masuda E, et al.
WDDD: Worm developmental dynamics database.
Nucleic Acids Research
41, D732-D737 (2013)
doi: 10.1093/nar/gks1107
Kimura A, Onami S.
Computer simulations and image processing reveal length-dependent pulling force as the primary mechanism for C. elegans male pronuclear migration.
Developmental Cell
8, 765-775 (2005)
doi: 10.1016/j.devcel.2005.03.007
ニュース

2025年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞、理研桜舞賞の授与について

2024年12月9日 研究成果
遺伝子同士の距離変化が発現を調節する新たな仕組みを発見
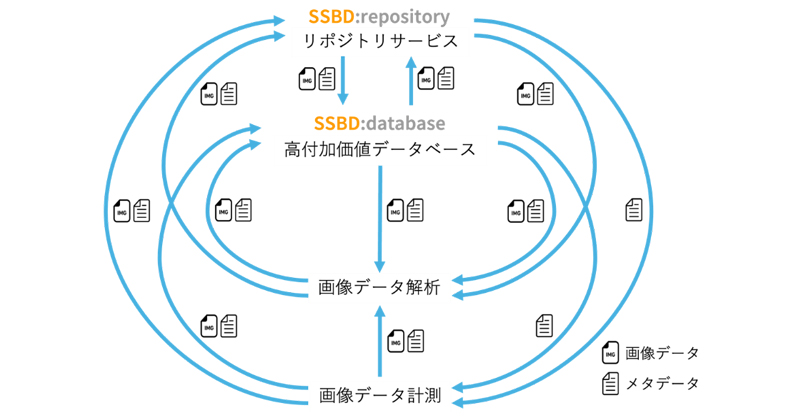
2024年10月31日 研究成果
画像データの共有がもたらす生命科学の発展
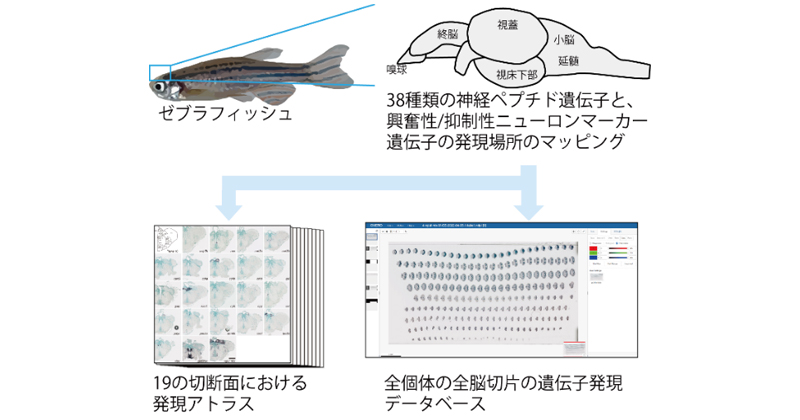
2024年6月4日 研究成果
ゼブラフィッシュ脳遺伝子発現データベースの作製・公開

2023年4月6日 研究成果
転写が盛んなゲノム領域も「塊」を作っていた!

2022年6月6日 研究成果
細胞が成長する過程におけるDNAの「ゆらぎ」をとらえた!
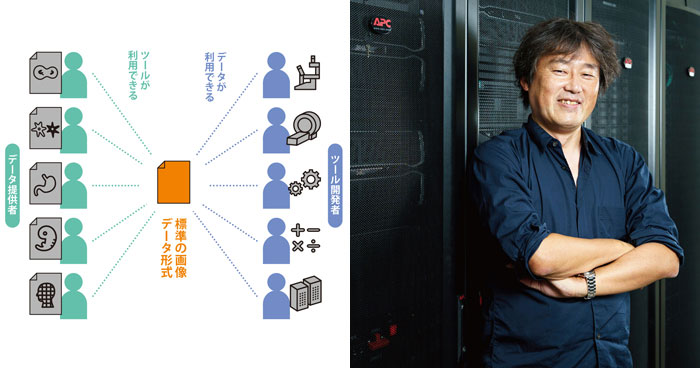
2021年10月26日 BDRニュース
研究最前線
データの「共有」で生命科学研究のレベルをジャンプアップ!

2021年5月25日 研究成果
“誕生日タグづけ”マウスの脳画像データベース「NeuroGT」を公開
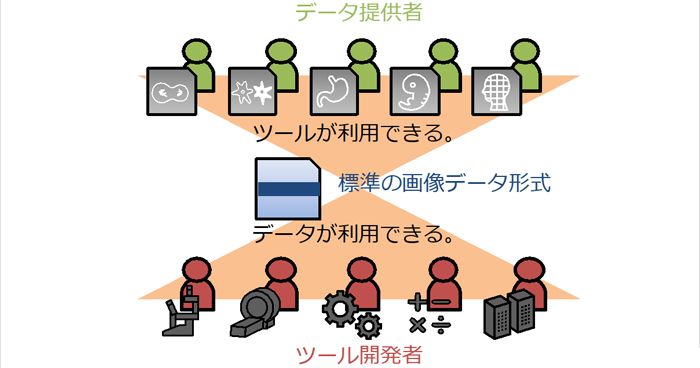
2021年5月11日 BDRニュース
生命科学画像のデータ形式の標準化とデータ共有リポジトリの整備に向けた国際提言

2020年9月25日 BDRニュース
新海創也研究員のエッセイが産経新聞の連載「科学の中身」に掲載されました
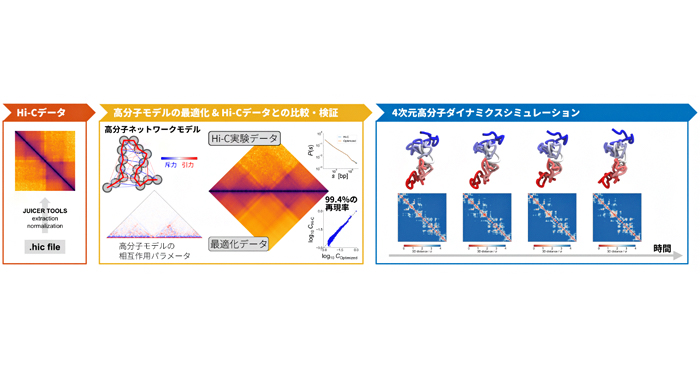
2020年8月7日 研究成果
ゲノムの動きをシミュレーションする新手法

2019年10月1日 BDRニュース
BDRの研究ネホリハホリ
データドリブン・ライフサイエンス






