チームディレクター
新津 藍
Ph.D.
生体分子動態デザイン研究チーム
[2025年4月よりBDRから生命医科学研究センター(IMS)に所属が変わりました]
拠点横浜/西研究棟
E-mailai.niitsu@riken.jp
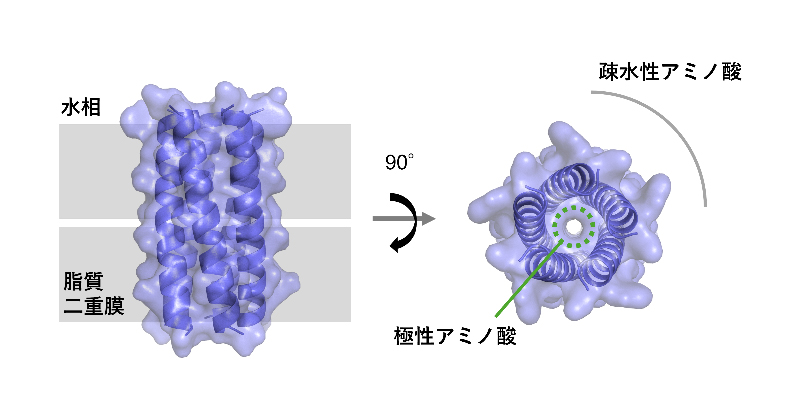
生体分子が機能する際には、その構造ダイナミクスが重要な鍵となります。生体分子動態デザイン研究チームでは、タンパク質デザインと分子シミュレーションの手法を組み合わせて、膜タンパク質・ペプチドを中心に構造ダイナミクスが制御可能な生体分子を設計する技術を確立し、設計分子の構造・機能解析を通して生体内で機能する仕組みを解明することを目指しています。また化学の視点を取り入れて設計分子を機能開拓することで、細胞動態の摂動・イメージングツール、バイオセンサーなどを創出し生命科学研究に役立てていきます。
主要論文
Tan C, Niitsu A, Sugita Y.
Highly Charged Proteins and Their Repulsive Interactions Antagonize Biomolecular Condensation.
Journal of American Chemical Society Au
3(3), 834-848 (2023)
doi: 10.1021/jacsau.2c00646
Niitsu A, Sugita Y.
Towards de novo design of transmembrane α-helical assemblies using structural modelling and molecular dynamics simulation.
Physical Chemistry Chemical Physics
25(5), 3595-3606 (2023)
doi: 10.1039/d2cp03972a
Scott AJ, Niitsu A, Kratochvil HT, et al.
Constructing ion channels from water-soluble α-helical barrels.
Nature Chemistry
13(7), 643-650 (2021)
doi: 10.1038/s41557-021-00688-0
Niitsu A, Re S, Oshima H, et al.
De Novo Prediction of Binders and Nonbinders for T4 Lysozyme by gREST Simulations.
Journal of Chemical Information and Modeling
59(9), 3879-3888 (2019)
doi: 10.1021/acs.jcim.9b00416
Niitsu A, Egawa A, Ikeda K, et al.
Veratridine binding to a transmembrane helix of sodium channel Nav1.4 determined by solid-state NMR.
Bioorganic & Medicinal Chemistry
26(21), 5644-5653 (2018)
doi: 10.1016/j.bmc.2018.10.012
Thomas F, Niitsu A, Oregioni A, et al.
Conformational Dynamics of Asparagine at Coiled-Coil Interfaces.
Biochemistry
56(50), 6544-6554 (2017)
doi: 10.1021/acs.biochem.7b00848
Niitsu A, Heal JW, Fauland K, et al.
Membrane-spanning α-helical barrels as tractable protein-design targets.
Philosophical Transactions of the Royal Society B
372(1726), 20160213 (2017)
doi: 10.1098/rstb.2016.0213
Mahendran KR, Niitsu A, Kong L, et al.
A monodisperse transmembrane α-helical peptide barrel.
Nature Chemistry
9(5), 411-419 (2017)
doi: 10.1038/nchem.2647