
本研究チームでは、理論的研究と実験的研究の双方から、分化や進化といった複数の時間・空間スケールを持つ生命ダイナミクスの理解を目指しています。具体的には、微生物の実験室進化系と網羅的な表現型・遺伝子型解析を組み合わせることにより、環境変化に対する応答・適応・進化という異なる時間スケールを持つダイナミクスの絡み合いを解析し、それらが安定に進むための原理を探求します。また、発生ダイナミクスの数理モデルを用いて、細胞間の相互作用がどのようにして安定かつ多様な状態からなる細胞社会を築くか、それをもたらす細胞内・細胞間のダイナミクスがどのような性質を持つかを解析します。
研究テーマ
- 数理モデルを用いた多細胞生物の発生・分化過程の解析
- 微生物の実験室進化実験と網羅的な表現型・遺伝子型解析による進化ダイナミクスの解明
- 網羅的表現型・遺伝子型解析のための解析アルゴリズム開発
主要論文
Uchida Y, Shigenobu S, Takeda H, et al.
Potential contribution of intrinsic developmental stability toward body plan conservation.
BMC Biology
20(1), 82 (2022)
doi: 10.1186/s12915-022-01276-5
Furusawa C, Tanabe K, Ishii C, et al.
Decoding gut microbiota by imaging analysis of fecal samples.
iScience
24(12), 103481 (2021)
doi: 10.1016/j.isci.2021.103481
Maeda T, Iwasawa J, Kotani H, et al.
High-throughput laboratory evolution reveals evolutionary constraints in Escherichia coli.
Nature Communications
11, 5970 (2020)
doi: 10.1038/s41467-020-19713-w
Furusawa C, Horinouchi T, Maeda T.
Toward prediction and control of antibiotic-resistance evolution.
Current Opinion in Biotechnology
54, 45-49 (2018)
doi: 10.1016/j.copbio.2018.01.026
Yoshida M, Reyes SG, Tsuda S, et al.
Time-programmable drug dosing allows the manipulation, suppression and reversal of antibiotic drug resistance in vitro.
Nature Communications
8, 15589 (2017)
doi: 10.1038/ncomms15589
Horinouchi T, Suzuki S, Hirasawa T, et al.
Phenotypic convergence in bacterial adaptive evolution to ethanol stress.
Bmc Evolutionary Biology
15, 180 (2015)
doi: 10.1186/s12862-015-0454-6
Furusawa C, Kaneko K.
Global relationships in fluctuation and response in adaptive evolution.
Journal of the Royal Society Interface
12(109), 20150482 (2015)
doi: 10.1098/rsif.2015.0482
Kaneko K, Furusawa C, Yomo T.
Universal Relationship in Gene-Expression Changes for Cells in Steady-Growth State.
Physical Review X
5(1), 011014 (2015)
doi: 10.1103/PhysRevX.5.011014
Suzuki S, Horinouchi T, Furusawa C.
Prediction of antibiotic resistance by gene expression profiles.
Nature Communications
5, 5792 (2014)
doi: 10.1038/ncomms6792
Horinouchi T, Minamoto T, Suzuki S, et al.
Development of an Automated Culture System for Laboratory Evolution.
Journal of Laboratory Automation
19(5), 478-482 (2014)
doi: 10.1177/2211068214521417
Ohno S, Shimizu H, Furusawa C.
FastPros: screening of reaction knockout strategies for metabolic engineering.
Bioinformatics
30(7), 981-987 (2014)
doi: 10.1093/bioinformatics/btt672
Furusawa C, Kaneko K.
A Dynamical-Systems View of Stem Cell Biology.
Science
338(6104), 215-217 (2012)
doi: 10.1126/science.1224311
Furusawa C, Kaneko K.
Adaptation to Optimal Cell Growth through Self-Organized Criticality.
Physical Review Letters
108(20), 208103 (2012)
doi: 10.1103/PhysRevLett.108.208103
ニュース

2025年10月10日 研究成果
細胞増殖法則を80年、180年越しに統一する原理を解明

2025年5月12日 研究成果
細菌のゲノム構造進化を実験室で観測
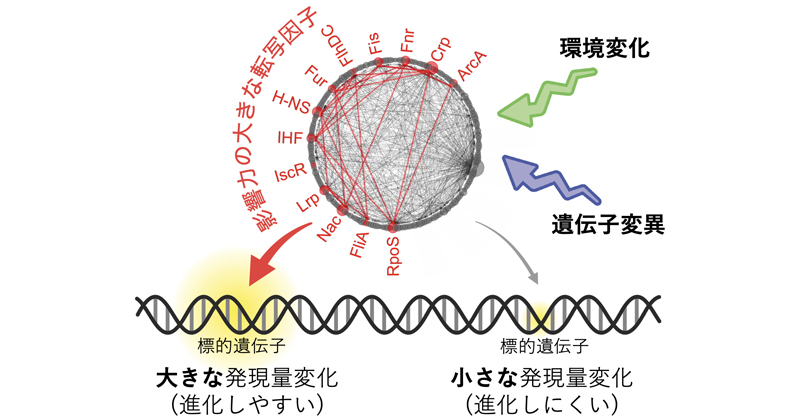
2025年3月21日 研究成果
発現量が進化しやすい遺伝子を細菌で発見

2024年5月13日 BDRニュース
BDRの研究ネホリハホリ
実験自動化に大事なこと

2022年12月14日 研究成果
微生物の薬剤耐性進化を大規模データから予測

2022年6月16日 BDRニュース
BDRの研究ネホリハホリ
進化と発生の揺らぎ?

2022年4月11日 研究成果
動物の形態進化は、「体づくり過程の変化しにくさ」に制限されてきた

2022年1月24日 研究成果
オペロン構造の進化過程の実証実験に成功
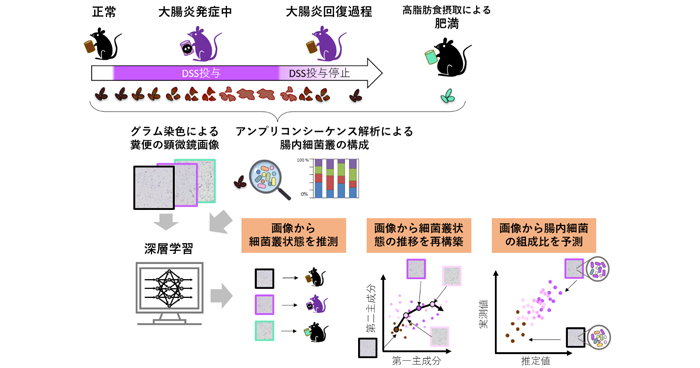
2021年12月2日 研究成果
腸内細菌叢の状態を細胞画像から読み解く

2021年2月8日 BDRニュース
研究者にズームイン
進化に失敗がある!?
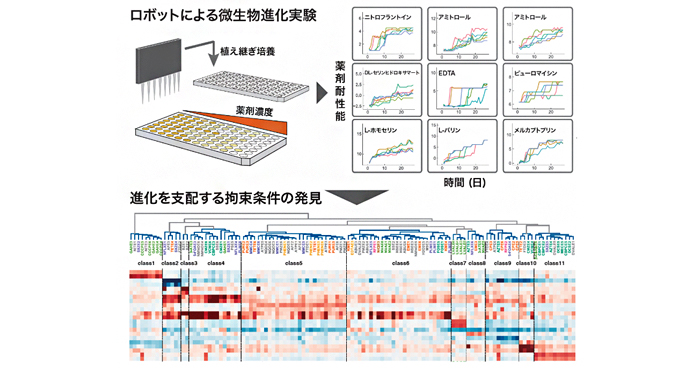
2020年11月24日 研究成果
ロボットによる微生物の大規模進化実験





