チームディレクター
平谷 伊智朗
Ph.D.
発生エピジェネティクス研究チーム
拠点 神戸/発生・再生研究棟
E-mail ichiro.hiratani[at]riken.jp
[at]を@に変えてください
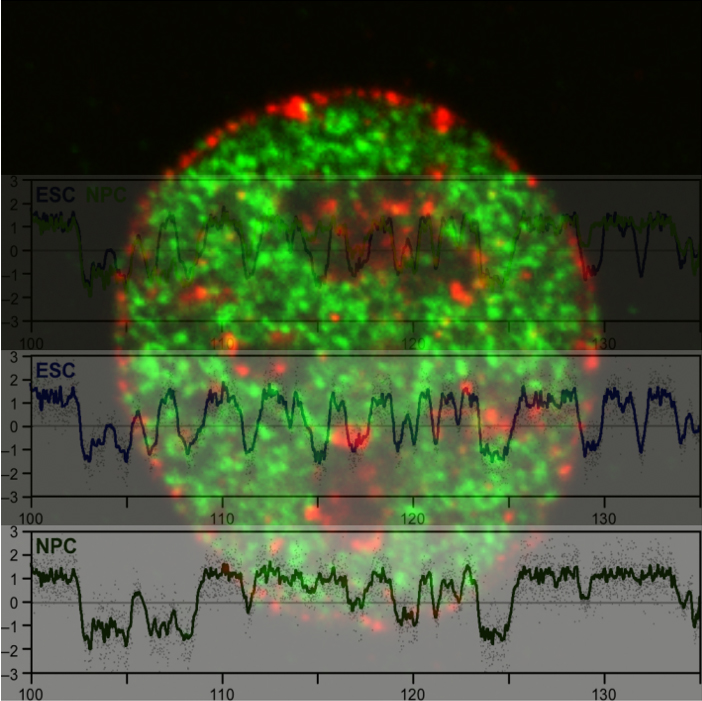
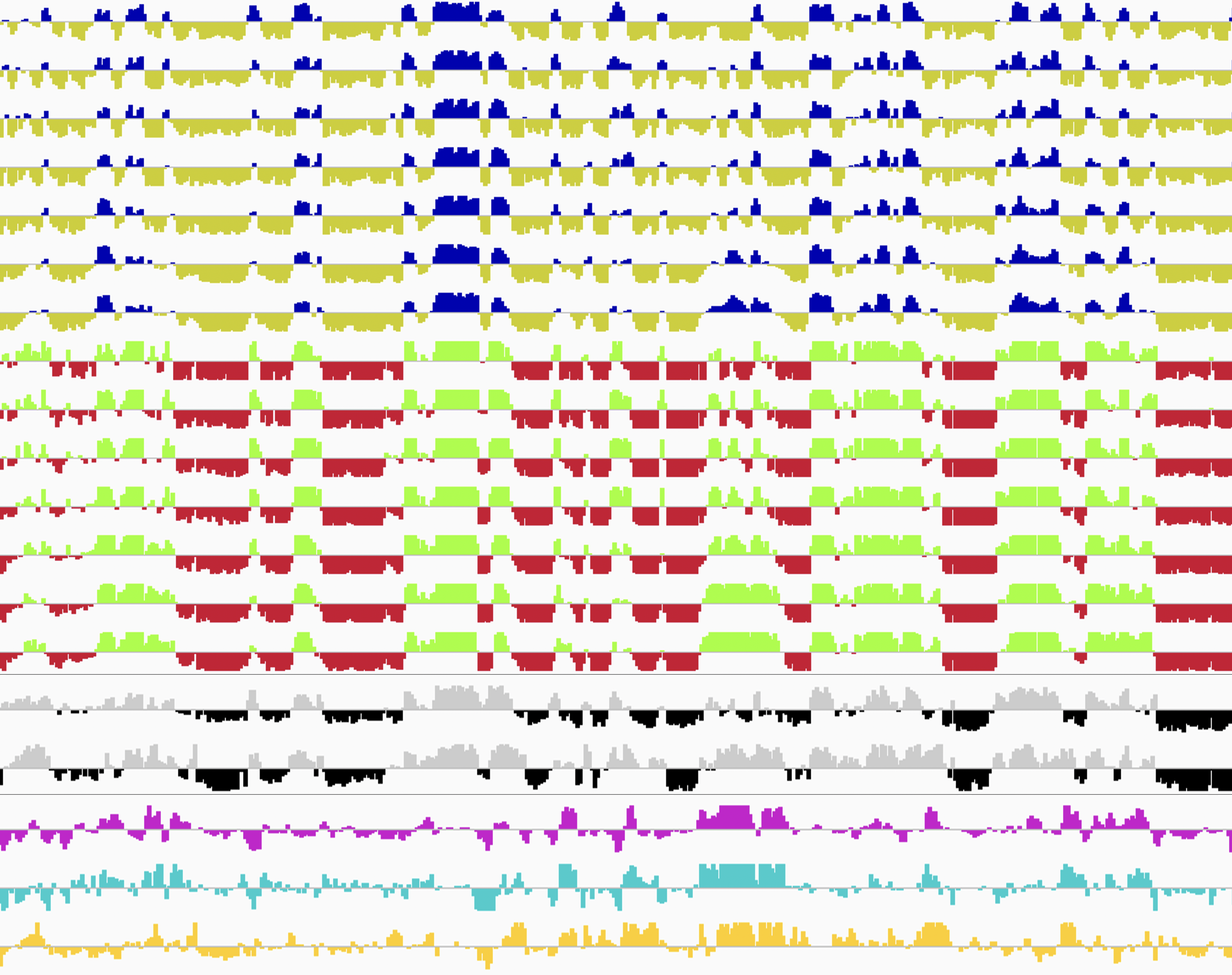
あらゆる生物の遺伝の本体であるゲノムDNAは、細胞核の中で三次元的に高度に折りたたまれています。ゲノムの三次元構造の制御を正しく理解することは、遺伝子発現をはじめ、様々なゲノム機能の基礎的な理解につながるので、生命科学全般にとって極めて重要ですが、未解明な問題が多く残されています。生き物を形づくる様々な細胞種に特異的なゲノム三次元構造はどのように形成・維持されているのでしょうか?そして、発生・分化・発達に伴って細胞の個性が変わるとゲノム三次元構造はどのように変化するのでしょうか?我々はゲノム三次元構造を良く反映することが知られているDNA複製制御を切り口に研究を展開しており、特に、独自に開発した1細胞全ゲノム解析技術であるscRepli-seq法と、ゲノム三次元構造の全ゲノム解析法Hi-Cなどの先端技術を上手く組み合わせて、マウス初期胚から培養細胞まで、様々な材料を用いて1細胞レベルで上記の難題に挑戦しています。
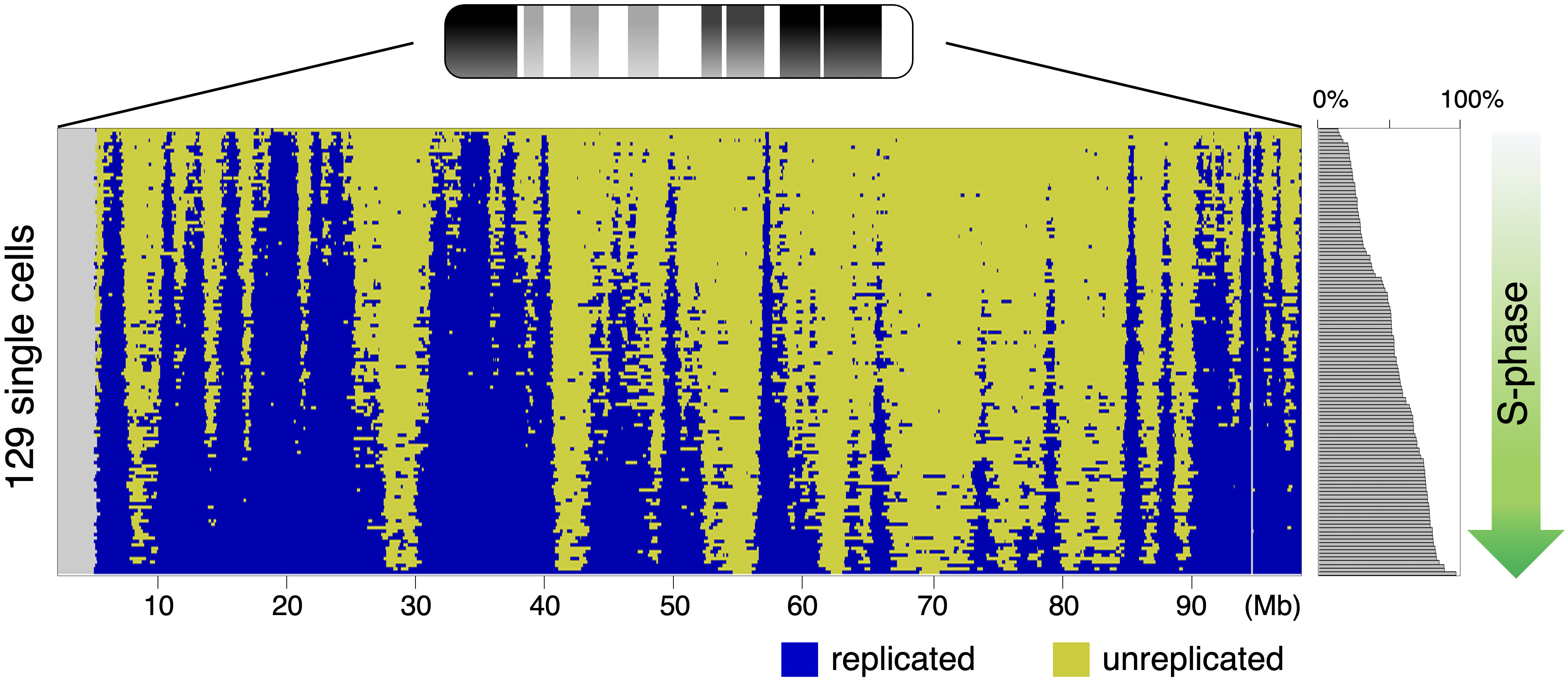
1細胞全ゲノムDNA複製解析(scRepli-seq)データ
研究テーマ
- ゲノム三次元構造とDNA複製の発生動態解析
- ゲノム三次元構造の制御機構の解析
- ゲノム三次元構造解明に向けた1細胞解析技術の開発
主要論文
Miura H, Hiratani I.
Cell cycle dynamics and developmental dynamics of the 3D genome: toward linking the two timescales.
Current Opinion in Genetics & Development
73, 101898 (2022)
doi: 10.1016/j.gde.2021.101898
Hada M, Miura H, Tanigawa A, et al.
Highly rigid H3.1/H3.2-H3K9me3 domains set a barrier for cell fate reprogramming in trophoblast stem cells.
Genes & Development
36, 84-102 (2022)
doi: 10.1101/gad.348782.121
Connolly C, Takahashi S, Miura H, et al.
SAF-A promotes origin licensing and replication fork progression to ensure robust DNA replication.
Journal of Cell Science
135(2), jcs258991 (2022)
doi: 10.1242/jcs.258991
Poonperm R, Hiratani I.
Formation of a multi-layered 3-dimensional structure of the heterochromatin compartment during early mammalian development.
Development, Growth & Differentiation
63(1), 5-17 (2021)
doi: 10.1111/dgd.12709
Miura H, Takahashi S, Shibata T, et al.
Mapping replication timing domains genome wide in single mammalian cells with single-cell DNA replication sequencing.
Nature Protocols
15(12), 4058-4100 (2020)
doi: 10.1038/s41596-020-0378-5
Kadota M, Nishimura O, Miura H, et al.
Multifaceted Hi-C benchmarking: what makes a difference in chromosome-scale genome scaffolding?
GigaScience
9(1), giz158 (2020)
doi: 10.1093/gigascience/giz158
Abdalla MOA, Yamamoto T, Maehara K, et al.
The Eleanor ncRNAs activate the topological domain of the ESR1 locus to balance against apoptosis.
Nature Communications
10, 3778 (2019)
doi: 10.1038/s41467-019-11378-4
Miura H, Takahashi S, Poonperm R, et al.
Single-cell DNA replication profiling identifies spatiotemporal developmental dynamics of chromosome organization.
Nature Genetics
51(9), 1356-1368 (2019)
doi: 10.1038/s41588-019-0474-z
Hiratani I, Takahashi S.
DNA Replication Timing Enters the Single-Cell Era.
Genes
10(3), 221 (2019)
doi: 10.3390/genes10030221
Takahashi S, Miura H, Shibata T, et al.
Genome-wide stability of the DNA replication program in single mammalian cells.
Nature Genetics
51(3), 529-540 (2019)
doi: 10.1038/s41588-019-0347-5
メンバー
平谷 伊智朗
チームディレクター
三浦 尚
上級研究員
POONPERM Rawin
研究員
髙橋 沙央里
研究員
大字 亜沙美
基礎科学特別研究員
谷川 明恵
テクニカルスタッフⅠ
一ノ瀨 孝子
テクニカルスタッフⅠ
ELUMALAI Jothivanan
研修生
CHOUBANI Linda Jade
国際プログラム・アソシエイト
ニュース

2025年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞、理研桜舞賞の授与について

2025年1月23日 BDRニュース
平谷伊智朗チームリーダーが兵庫県科学賞を受賞
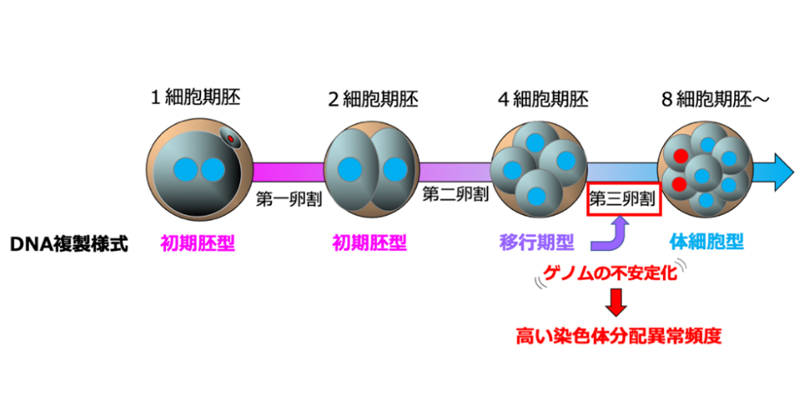
2024年8月29日 研究成果
マウス初期胚の型破りなDNA複製様式を発見

2024年8月8日 BDRニュース
一般向けイベント
高校生のための生命科学体験講座2024を開催しました

2024年4月15日 BDRニュース
研究人十色
国境も研究分野も越えて、自分らしく研究する

2024年3月14日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について
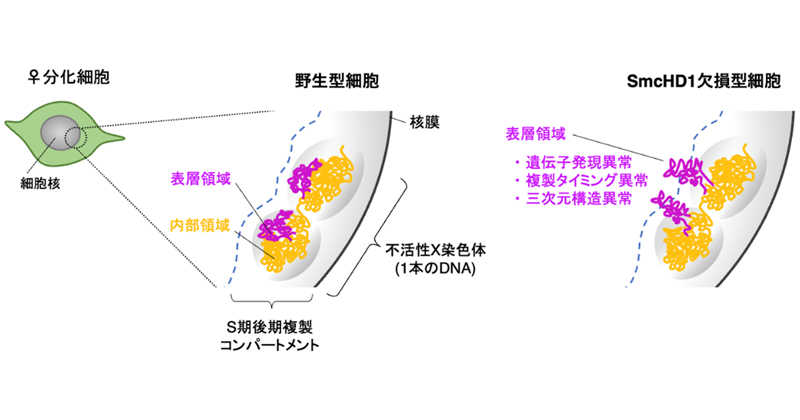
2023年8月11日 研究成果
X染色体不活化の安定性は染色体の形が鍵だった
2023年2月9日 BDRニュース
平谷チームリーダーが井上学術賞を受賞
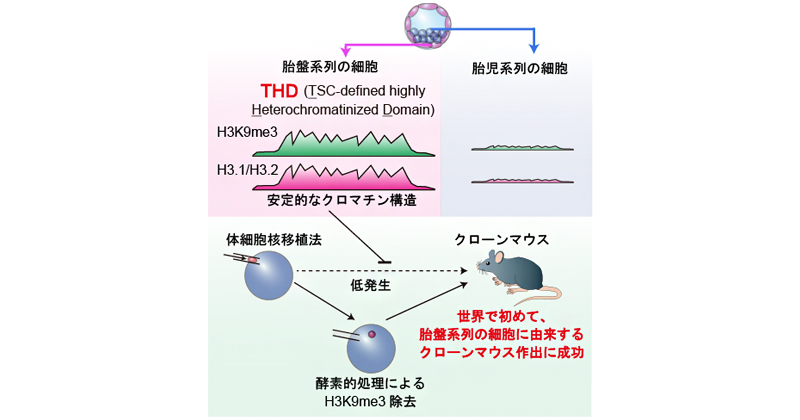
2022年1月18日 研究成果
胎盤らしさを支える分子基盤を解明
2021年6月3日 BDRニュース
BDRの研究ネホリハホリ
ドキドキ!スクリーニング!
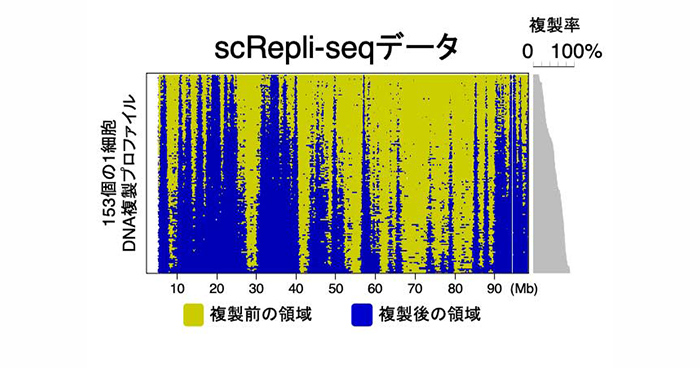
2020年11月24日 研究成果
インフォマティクス初心者でも1細胞全ゲノムDNA解析が可能に

2019年12月9日 BDRニュース
理研ニュース12月号に平谷伊智朗チームリーダー(発生エピジェネティクス研究チーム)らの研究が紹介されました

2019年8月22日 研究成果
ゲノムDNAの立体構造から見えた乳がん細胞の弱点
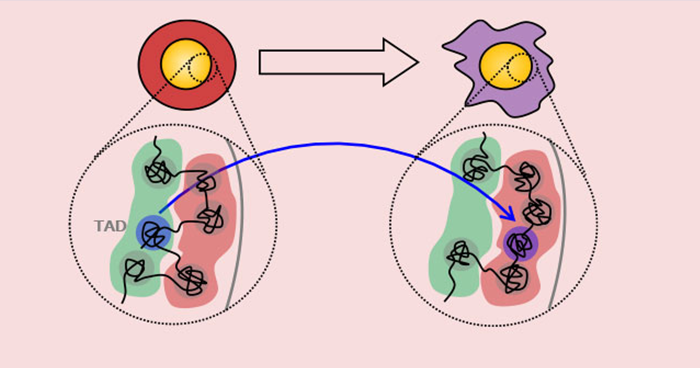
2019年8月13日 研究成果
染色体の形は細胞分化と共にこう変わる

2019年6月12日 BDRニュース
2018年度「理研栄峰賞」および2018年度「理研梅峰賞」の受賞者が決まりました

2019年2月26日 研究成果
ゲノムDNA複製の真の姿を捉えた