
生命は高分子、細胞、臓器などあらゆるレベルで我々人類の工学的産物をはるかに凌駕する緻密さを持っています。例えば、ヒトの脳は10MWを消費するスーパーコンピュータに比べおよそ百万倍効率的です。我々は、人工知能やシミュレーションといった情報技術に生命の仕組みに触発された原理を取り入れ新しいパラダイムを生み出すこと、そして、最新の情報技術を科学研究の現場で応用すること、の二つの双対する問題設定を軸に様々な研究に取り組んでいます。 具体的には、①細胞シミュレーション技術の開発。特にゲノム情報からの自動モデル生成や1分子粒度の離散事象シミュレーション技術、またソフトウエア基盤E-Cellの開発。②ロボットと人工知能による実験自動化技術の開発。特にiPS細胞等の培養と分化誘導の自動化や細胞状態の推定と制御。また、③神経科学や細胞内反応ネットワークに触発された新規の情報処理手法の開発。特に全脳アーキテクチャの開発や高性能脳型人工知能基盤ソフトウエアBriCAの開発などに取り組んでいます。 我々の最終的な野望は、これらの技術を総合して、生命科学研究におけるあらゆるプロセスを自動化すること、つまり人工知能駆動型科学の姿を示すことです。
研究テーマ
- ゲノムスケール細胞シミュレーションソフトウエア基盤
- AIとロボットによる生命科学実験の自動化
- 脳型人工知能
主要論文
Kanda GN, Tsuzuki T, Terada M, et al.
Robotic search for optimal cell culture in regenerative medicine.
eLife
11, e77007 (2022)
doi: 10.7554/eLife.77007
Ochiai K, Motozawa N, Terada M, et al.
A Variable Scheduling Maintenance Culture Platform for Mammalian Cells.
SLAS technology
26(2), 209-217 (2021)
doi: 10.1177/2472630320972109
Watabe M, Yoshimura H, Arjunan SNV, et al.
Signaling activations through G-protein-coupled-receptor aggregations.
Phys Rev E.
102, 032413 (2020)
doi: 10.1103/PhysRevE.102.032413
Yachie N, Natsume T, Takahashi K, et al.
Robotic crowd biology with Maholo LabDroids.
Nature Biotechnology
35(4), 310-312 (2017)
doi: 10.1038/nbt.3758
Yamakawa H, Arakawa N, Takahashi K, et al.
Reinterpreting The Cortical Circuit
Pre-proceedings of the IJCAI-17 Workshop on Architectures for Generality & Autonomy
(2017)
Iwamoto K, Shindo Y, Takahashi K.
Modeling Cellular Noise Underlying Heterogeneous Cell Responses in the Epidermal Growth Factor Signaling Pathway.
Plos Computational Biology
12(11), e1005222 (2016)
doi: 10.1371/journal.pcbi.1005222
Itaya K, Takahashi K, Nakamura M, et al.
BriCA: A modular software platform for whole brain architecture
Neural information processing – 23rd international conference, ICONIP
(2016)
Shindo Y, Iwamoto K, Mouri K, et al.
Conversion of graded phosphorylation into switch-like nuclear translocation via autoregulatory mechanisms in ERK signalling.
Nature Communications
7, 10485 (2016)
doi: 10.1038/ncomms10485
Karr JR, Takahashi K, Funahashi A.
The principles of whole-cell modeling.
Current Opinion in Microbiology
27, 18-24 (2015)
doi: 10.1016/j.mib.2015.06.004
Watabe M, Arjunan SNV, Fukushima S, et al.
A Computational Framework for Bioimaging Simulation.
Plos One
10(7), e0130089 (2015)
doi: 10.1371/journal.pone.0130089
Shimo H, Arjunan SNV, Machiyama H, et al.
Particle Simulation of Oxidation Induced Band 3 Clustering in Human Erythrocytes.
Plos Computational Biology
11(6), UNSP e1004 (2015)
doi: 10.1371/journal.pcbi.1004210
Kaizu K, de Ronde W, Paijmans J, et al.
The Berg-Purcell Limit Revisited.
Biophysical Journal
106(4), 976-985 (2014)
doi: 10.1016/j.bpj.2013.12.030
Hihara S, Pack CG, Kaizu K, et al.
Local Nucleosome Dynamics Facilitate Chromatin Accessibility in Living Mammalian Cells.
Cell Reports
2(6), 1645-1656 (2012)
doi: 10.1016/j.celrep.2012.11.008
ニュース

2025年8月19日 研究成果
AIとロボットが実験の「裏方作業」もする未来

2025年5月9日 BDRニュース
BDRの研究ネホリハホリ
自動化のその先へ

2025年3月31日 研究成果
生きた細胞内で「ユークロマチン」と「ヘテロクロマチン」のふるまいを見分ける新技術を開発

2024年10月8日 BDRニュース
一般向けイベント
理化学研究所 科学講演会2024~研究者の“わくわく“が未来を紡ぐ~

2024年2月20日 研究成果
システム生物学のためのメタバース活用法
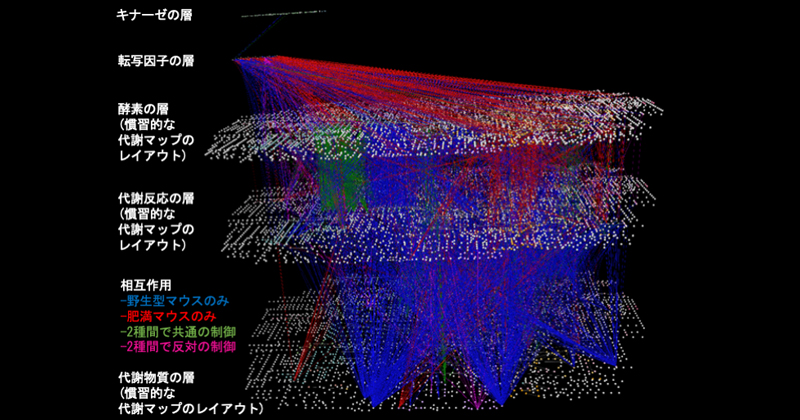
2024年2月19日 研究成果
細胞内の多階層ネットワークの可視化技術を開発

2024年1月5日 BDRニュース
中学生が聞きました!
研究者への道のり〜田中信行さんの場合〜

2023年12月25日 研究成果
周りを見て考えて手を動かす自動実験ロボ
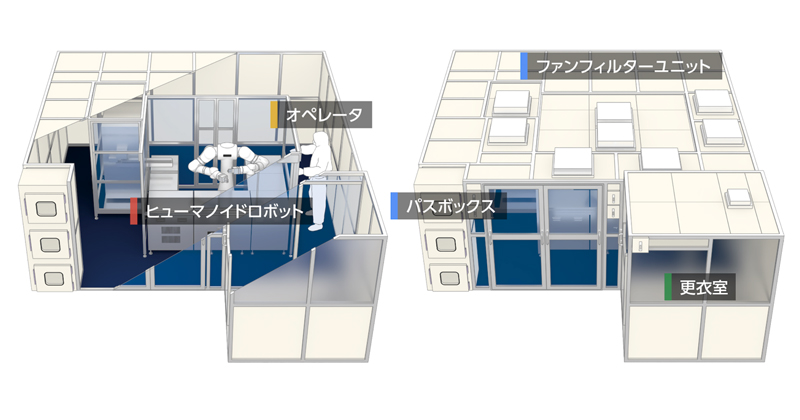
2023年11月30日 研究成果
ヒューマノイドロボットは再生医療の現場へ

2023年3月23日 BDRニュース
理研栄峰賞、理研梅峰賞の授与について

2022年11月4日 BDRニュース
研究最前線
DXとAIが新しい科学の世界を切り開く

2022年6月28日 研究成果
再生医療用細胞レシピをロボットとAIが自律的に試行錯誤

2022年2月14日 BDRニュース
研究者にズームイン
ヒューマノイドロボットがライフサイエンス研究をクリエイティブに躍進させる

2021年6月28日 研究成果
生命科学実験の効率的な自動化を実現するスケジューリング手法を開発

2021年1月22日 BDRニュース
西田孝三テクニカルスタッフのエッセイが産経新聞の連載「科学の中身」に掲載されました

2020年12月4日 研究成果
ヒューマノイドロボットとAIによる自律細胞培養





